機械学習のお勉強(データの前処理)①
教科書
データセットの欠損値の削除と補完
pandasを使ってデータを読み込むと欠損場所にNanが入る。 欠損値のカウントを行うこともでき、numpyにはvaluesによってアクセスできる。
import pandas as pd from io import StringIO csv_data = '''A,B,C,D 1,2,3,4 5,6,,8 10,11,12,''' csv_data = unicode(csv_data) df = pd.read_csv(StringIO(csv_data)) print df print df.values print df.isnull().sum()
削除
print df.dropna() print df.dropna(axis=1)
補完
from sklearn.preprocessing import Imputer imr = Imputer(missing_values='NaN', strategy='mean',axis=0) imr = imr.fit(df) imputed_data = imr.transform(df.values) imputed_data
機械学習のアルゴリズムに合わせたカテゴリデータの整形
順序特徴量のマッピング
df = pd.DataFrame([
['green', 'M',10.1,'class1'],
['red','L',13.5,'class2'],
['blue','XL',15.3,'class1']])
df.columns = ['color' , 'size', 'price', 'classlabel']
size_mapping ={'XL':3, 'L':2,'M':1}
df['size'] = df['size'].map(size_mapping)
print df
クラスラベルのエンコーディング
import numpy as np class_mapping = {label:idx for idx,label in enumerate(np.unique(df['classlabel']))} print class_mapping df['classlabel'] = df['classlabel'].map(class_mapping) print df inv_class_mapping = {v: k for k,v in class_mapping.items()} df['classlabel'] = df['classlabel'].map(inv_class_mapping) print df
Label Encoder
from sklearn.preprocessing import LabelEncoder class_le = LabelEncoder() y = class_le.fit_transform(df['classlabel'].values) print y print class_le.inverse_transform(y)
one-hotエンコーディング
X = df[['color', 'size', 'price']].values print X color_le = LabelEncoder() X[:, 0] = color_le.fit_transform(X[:, 0]) print X from sklearn.preprocessing import OneHotEncoder ohe = OneHotEncoder(categorical_features=[0]) print ohe.fit_transform(X).toarray() print pd.get_dummies(df[['price', 'color', 'size']])
データセットの分割
df_wine = pd.read_csv('https://raw.githubusercontent.com/rasbt/python-machine-learning-book/master/code/datasets/wine/wine.data', header=None) df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash', 'Alcalinity of ash', 'Magnesium', 'Total phenols', 'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins', 'Color intensity', 'Hue', 'OD280/OD315 of diluted wines', 'Proline'] df_wine.head() if Version(sklearn_version) < '0.18': from sklearn.cross_validation import train_test_split else: from sklearn.model_selection import train_test_split X, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values X_train, X_test, y_train, y_test = \ train_test_split(X, y, test_size=0.3, random_state=0)
標準化・正規化
正規化 0 ~ 1
from sklearn.preprocessing import MinMaxScaler mms = MinMaxScaler() X_train_norm = mms.fit_transform(X_train) # fit and trans X_test_norm = mms.transform(X_test) # trans only
標準化 平均:0 , 標準偏差:1
from sklearn.preprocessing import StandardScaler stdsc = StandardScaler() X_train_std = stdsc.fit_transform(X_train) X_test_std = stdsc.transform(X_test)
example
ex = pd.DataFrame([0, 1, 2, 3, 4, 5]) # standardize ex[1] = (ex[0] - ex[0].mean()) / ex[0].std(ddof=0) # Please note that pandas uses ddof=1 (sample standard deviation) # by default, whereas NumPy's std method and the StandardScaler # uses ddof=0 (population standard deviation) # normalize ex[2] = (ex[0] - ex[0].min()) / (ex[0].max() - ex[0].min()) ex.columns = ['input', 'standardized', 'normalized'] print ex
モデルの構築に適した特徴量の選択
汎化誤差を減らすための方法 1. 多くのデータを集める
正規化を通じて複雑さを減らす
パラメータの少ないシンプルなモデルを用いる
データの次元数を減らす
特徴量の選択:L1正規化
L2
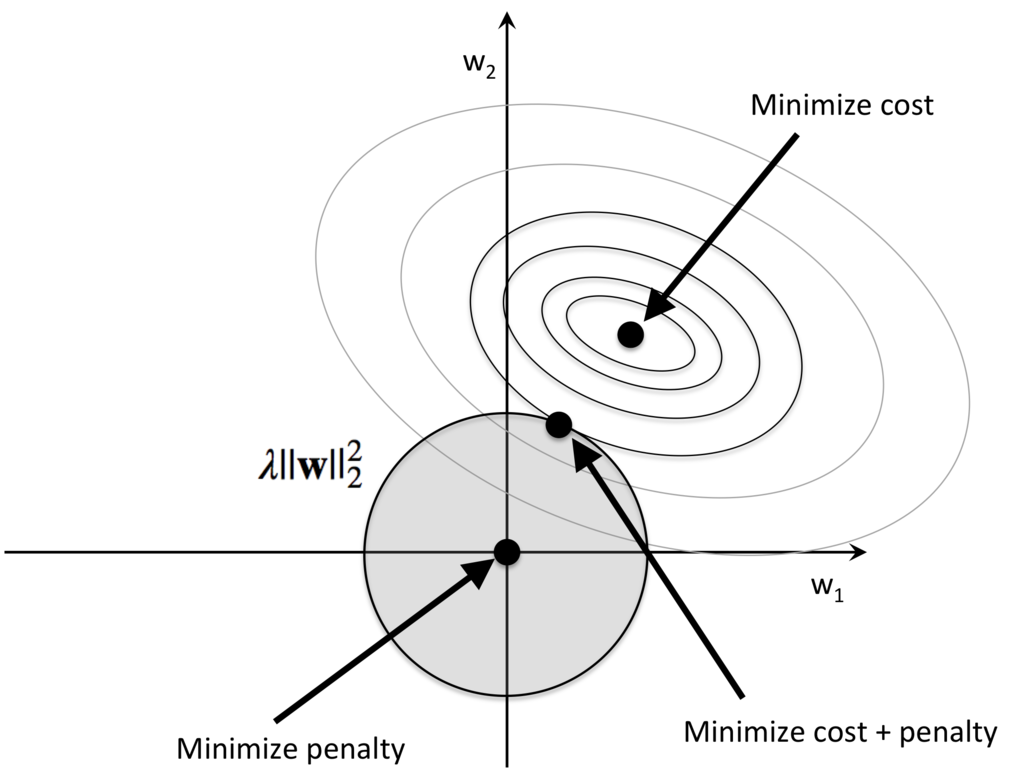
L1

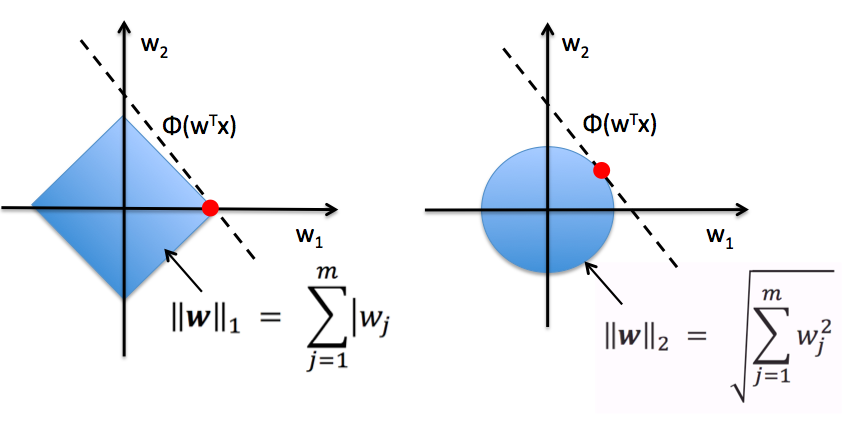
L1正規化は重みが軸上に乗ることが多く、疎な解が求まりやすく、特徴量の選択に有用。
無関係の特徴量が多い高次元のデータセットなどでの学習の場合、汎化性能の良い結果が得られる。
from sklearn.linear_model import LogisticRegression lr = LogisticRegression(penalty='l1', C=0.1) lr.fit(X_train_std, y_train) print('Training accuracy:', lr.score(X_train_std, y_train)) print('Test accuracy:', lr.score(X_test_std, y_test)) print lr.coef_
正規化の強さに対する特徴量の重み
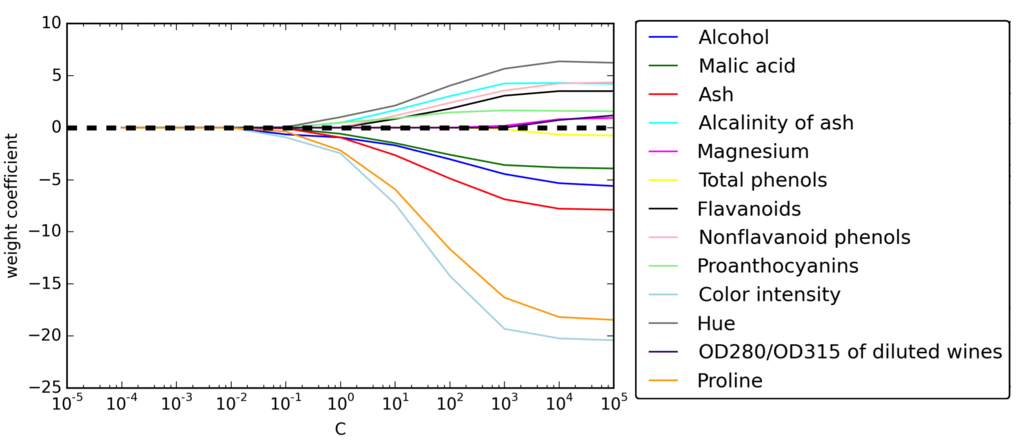
L1正規化のペナルティを増やすと特徴量がすべて0になる
次元削減(特徴選択・特徴抽出):特徴選択
フィルタ法、ラッパー法、埋め込み法の3つに大別される。
フィルタ法:情報利得やGini係数などを用いて、学習を伴わずに特徴量の重要性を測定して、有効な特徴量を選択する手法
ラッパー法:学習を行いながら重要な特徴量を選択する手法
埋め込み法:学習アルゴリズムに特徴量の選択も埋め込む。L1正規化、LASSOなどが該当。
逐次特徴選択アルゴリズム
目的
問題に最も関連がある特徴量の部分集合を自動的に選択する
無関係の特徴量やノイズを取り除くことで、モデルの汎化誤差を削減する
逐次後退選択
d次元の特徴空間をk次元の特徴部分空間に削除する
アルゴリズムをk=dで初期化
Jの評価を最大化する特徴量x^を決定する。x^ = argmax J(Xk - x) 。 x ∈Xk 。
特徴量の集合から特徴量x^を削除する
kが目的とする特徴量の個数に等しくなれば終了。そうでなければstep2へ。
実装
from sklearn.base import clone from itertools import combinations import numpy as np from sklearn.metrics import accuracy_score if Version(sklearn_version) < '0.18': from sklearn.cross_validation import train_test_split else: from sklearn.model_selection import train_test_split class SBS(): def __init__(self, estimator, k_features, scoring=accuracy_score, test_size=0.25, random_state=1): self.scoring = scoring self.estimator = clone(estimator) self.k_features = k_features self.test_size = test_size self.random_state = random_state def fit(self, X, y): X_train, X_test, y_train, y_test = \ train_test_split(X, y, test_size=self.test_size, random_state=self.random_state) dim = X_train.shape[1] self.indices_ = tuple(range(dim)) self.subsets_ = [self.indices_] score = self._calc_score(X_train, y_train, X_test, y_test, self.indices_) self.scores_ = [score] while dim > self.k_features: scores = [] subsets = [] for p in combinations(self.indices_, r=dim - 1): score = self._calc_score(X_train, y_train, X_test, y_test, p) scores.append(score) subsets.append(p) best = np.argmax(scores) self.indices_ = subsets[best] self.subsets_.append(self.indices_) dim -= 1 self.scores_.append(scores[best]) self.k_score_ = self.scores_[-1] return self def transform(self, X): return X[:, self.indices_] def _calc_score(self, X_train, y_train, X_test, y_test, indices): self.estimator.fit(X_train[:, indices], y_train) y_pred = self.estimator.predict(X_test[:, indices]) score = self.scoring(y_test, y_pred) return score
KNNで実装
import matplotlib.pyplot as plt from sklearn.neighbors import KNeighborsClassifier knn = KNeighborsClassifier(n_neighbors=2) # selecting features sbs = SBS(knn, k_features=1) sbs.fit(X_train_std, y_train) # plotting performance of feature subsets k_feat = [len(k) for k in sbs.subsets_] plt.plot(k_feat, sbs.scores_, marker='o') plt.ylim([0.7, 1.1]) plt.ylabel('Accuracy') plt.xlabel('Number of features') plt.grid() plt.tight_layout() # plt.savefig('./sbs.png', dpi=300) plt.show()
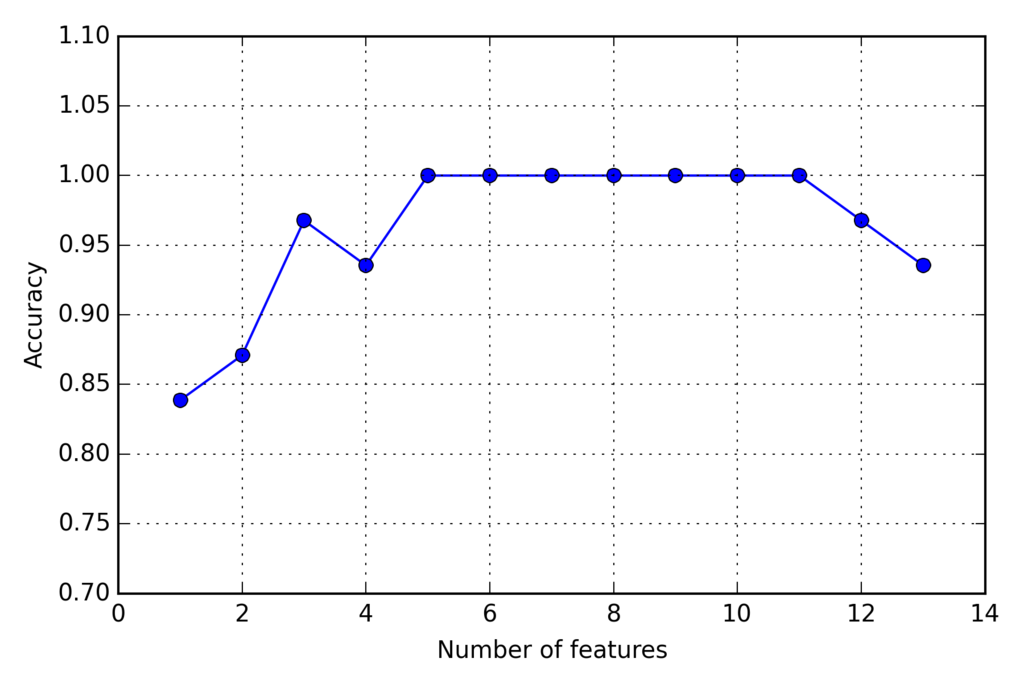 次元削減されたk=5で分類器が100%の正解率を達成している
次元削減されたk=5で分類器が100%の正解率を達成している
選ばれた特徴量は
k5 = list(sbs.subsets_[8]) print(df_wine.columns[1:][k5])
以下の5つ
(['Alcohol', 'Malic acid', 'Alcalinity of ash', 'Hue', 'Proline'], dtype='object')
次元削減前
knn.fit(X_train_std, y_train) print('Training accuracy:', knn.score(X_train_std, y_train)) print('Test accuracy:', knn.score(X_test_std, y_test))
結果
Training accuracy: 0.983870967742 Test accuracy: 0.944444444444
テストでの結果減。若干の過学習の傾向。
次元削減後
knn.fit(X_train_std[:, k5], y_train) print('Training accuracy:', knn.score(X_train_std[:, k5], y_train)) print('Test accuracy:', knn.score(X_test_std[:, k5], y_test))
結果
Training accuracy: 0.959677419355 Test accuracy: 0.962962962963
テストでの結果向上。過学習が軽減。
ランダムフォレストで特徴量の重要度測定
線形分離可能かどうかについて、前提を設けなくても、フォレスト内のすべての決定木から計算された不純度の平均的な減少量として特徴量の重要度を測定できる。
Winwデータセットの13個の特徴量の重要度に基づいてランク付け
from sklearn.ensemble import RandomForestClassifier feat_labels = df_wine.columns[1:] forest = RandomForestClassifier(n_estimators=10000, random_state=0, n_jobs=-1) forest.fit(X_train, y_train) importances = forest.feature_importances_ indices = np.argsort(importances)[::-1] for f in range(X_train.shape[1]): print("%2d) %-*s %f" % (f + 1, 30, feat_labels[indices[f]], importances[indices[f]])) plt.title('Feature Importances') plt.bar(range(X_train.shape[1]), importances[indices], color='lightblue', align='center') plt.xticks(range(X_train.shape[1]), feat_labels[indices], rotation=90) plt.xlim([-1, X_train.shape[1]]) plt.tight_layout() #plt.savefig('./random_forest.png', dpi=300) plt.show()
重要度の総和が0になるように正規化されている。

閾値で重要度の高い特徴量の抽出
if Version(sklearn_version) < '0.18': X_selected = forest.transform(X_train, threshold=0.15) else: from sklearn.feature_selection import SelectFromModel sfm = SelectFromModel(forest, threshold=0.15, prefit=True) X_selected = sfm.transform(X_train) print X_selected.shape for f in range(X_selected.shape[1]): print("%2d) %-*s %f" % (f + 1, 30, feat_labels[indices[f]], importances[indices[f]]))
重要度の高い3つが選ばれる
1) Color intensity 0.182483 2) Proline 0.158610 3) Flavanoids 0.150948