機械学習のお勉強(pytorchのtutorialを眺めてみる)

install
DOCS
PyTorch documentation — PyTorch master documentation
Tutorial
すごくわかりやすい
What is PyTorch? — PyTorch Tutorials 0.2.0_4 documentation
Pytorchで遊ぼう【データ成形からFNNまで】 - HELLO CYBERNETICS
GitHub - yunjey/pytorch-tutorial: PyTorch Tutorial for Deep Learning Researchers
気になった箇所だけ下にまとめる👇
Data Loading and Processing Tutorial
Data Loading and Processing Tutorial — PyTorch Tutorials 0.2.0_4 documentation
Transform
Let’s create three transforms:
Rescale: to scale the image
RandomCrop: to crop from image randomly. This is data augmentation.
ToTensor: to convert the numpy images to torch images (we need to swap axes).
Iterating through the dataset
Batching the data
Shuffling the data
Load the data in parallel using multiprocessing workers
num_workersでいくつのコアでデータをロードするか指定(デフォルトはメインのみ)
dataloader = DataLoader(transformed_dataset, batch_size=4, shuffle=True, num_workers=4) # Helper function to show a batch def show_landmarks_batch(sample_batched): """Show image with landmarks for a batch of samples.""" images_batch, landmarks_batch = \ sample_batched['image'], sample_batched['landmarks'] batch_size = len(images_batch) im_size = images_batch.size(2) grid = utils.make_grid(images_batch) plt.imshow(grid.numpy().transpose((1, 2, 0))) for i in range(batch_size): plt.scatter(landmarks_batch[i, :, 0].numpy() + i * im_size, landmarks_batch[i, :, 1].numpy(), s=10, marker='.', c='r') plt.title('Batch from dataloader') for i_batch, sample_batched in enumerate(dataloader): print(i_batch, sample_batched['image'].size(), sample_batched['landmarks'].size()) # observe 4th batch and stop. if i_batch == 3: plt.figure() show_landmarks_batch(sample_batched) plt.axis('off') plt.ioff() plt.show() break
output
0 torch.Size([4, 3, 224, 224]) torch.Size([4, 68, 2]) 1 torch.Size([4, 3, 224, 224]) torch.Size([4, 68, 2]) 2 torch.Size([4, 3, 224, 224]) torch.Size([4, 68, 2]) 3 torch.Size([4, 3, 224, 224]) torch.Size([4, 68, 2])
torchvision
これは便利すぎる
One of the more generic datasets available in torchvision is ImageFolder.
It assumes that images are organized in the following way:
root/ants/xxx.png
root/ants/xxy.jpeg
root/ants/xxz.png
.
.
.
root/bees/123.jpg
root/bees/nsdf3.png
root/bees/asd932_.png
where ‘ants’, ‘bees’ etc. are class labels. Similarly generic transforms which operate on PIL.Image like RandomHorizontalFlip, Scale, are also avaiable. You can use these to write a dataloader like this:
import torch from torchvision import transforms, datasets data_transform = transforms.Compose([ transforms.RandomSizedCrop(224), transforms.RandomHorizontalFlip(), transforms.ToTensor(), transforms.Normalize(mean=[0.485, 0.456, 0.406], std=[0.229, 0.224, 0.225]) ]) hymenoptera_dataset = datasets.ImageFolder(root='hymenoptera_data/train', transform=data_transform) dataset_loader = torch.utils.data.DataLoader(hymenoptera_dataset, batch_size=4, shuffle=True, num_workers=4)
これを使えば自作データセットのCNNのデータの用意がすぐできるイエイ
Transfer Learning Tutorial
Transfer Learning tutorial — PyTorch Tutorials 0.2.0_4 documentation
These two major transfer learning scenarios looks as follows:
Finetuning the convnet: Instead of random initializaion, we initialize the network with a pretrained network, like the one that is trained on imagenet 1000 dataset. Rest of the training looks as usual.
ConvNet as fixed feature extractor: Here, we will freeze the weights for all of the network except that of the final fully connected layer. This last fully connected layer is replaced with a new one with random weights and only this layer is trained.
Finetuning the convnet
pretrainされたモデルを読み込んだ上で学習
model_ft = models.resnet18(pretrained=True) num_ftrs = model_ft.fc.in_features model_ft.fc = nn.Linear(num_ftrs, 2) if use_gpu: model_ft = model_ft.cuda() criterion = nn.CrossEntropyLoss() # Observe that all parameters are being optimized optimizer_ft = optim.SGD(model_ft.parameters(), lr=0.001, momentum=0.9) # Decay LR by a factor of 0.1 every 7 epochs exp_lr_scheduler = lr_scheduler.StepLR(optimizer_ft, step_size=7, gamma=0.1)
ConvNet as fixed feature extractor
for param in model_conv.parameters(): param.requires_grad = False
ここでconv層の重みは固定をして全結合層だけ学習
model_conv = torchvision.models.resnet18(pretrained=True) for param in model_conv.parameters(): param.requires_grad = False # Parameters of newly constructed modules have requires_grad=True by default num_ftrs = model_conv.fc.in_features model_conv.fc = nn.Linear(num_ftrs, 2) if use_gpu: model_conv = model_conv.cuda() criterion = nn.CrossEntropyLoss() # Observe that only parameters of final layer are being optimized as # opoosed to before. optimizer_conv = optim.SGD(model_conv.fc.parameters(), lr=0.001, momentum=0.9) # Decay LR by a factor of 0.1 every 7 epochs exp_lr_scheduler = lr_scheduler.StepLR(optimizer_conv, step_size=7, gamma=0.1)
GPU化
Training a classifier — PyTorch Tutorials 0.2.0_4 documentation
Multi-GPU examples — PyTorch Tutorials 0.2.0_4 documentation
net.cuda() inputs, labels = Variable(inputs.cuda()), Variable(labels.cuda())
上記のように基本的にinputとラベルをすべてcuda()とつけてgpu化する。
.cpu()とするとcpu化。
pytorchのdebianファイル
GitHub - CDLuminate/pytorch: PyTorch Debian packaging
参考
pytorch.learning/mnist1.py at master · moskomule/pytorch.learning · GitHub
Inferring shape via flatten operator - PyTorch Forums
ChainerとPyTorchのコードを比較する - 線形回帰編 - Qiita
PyTorch : Tutorial 初級 : 分類器を訓練する – CIFAR-10 – PyTorch
ssh上でのmatplotlibのエラー
機械学習のお勉強(pytorchを使ってみる)

すごくわかりやすい参考、講義
fast.ai · Making neural nets uncool again
Practical Deep Learning with PyTorch | Udemy
PyTorch – Pytorch MXNet Caffe2 ドキュメント/応用 – クラスキャット
Practical Deep Learning with PyTorch | Udemy
インストール
ソースから GitHub - pytorch/pytorch: Tensors and Dynamic neural networks in Python with strong GPU acceleration
Tutorial
GitHub - yunjey/pytorch-tutorial: PyTorch Tutorial for Deep Learning Researchers
CNN参考
PyTorch-Mini-Tutorials/5_convolutional_net.py at master · vinhkhuc/PyTorch-Mini-Tutorials · GitHub
PyTorch-Tutorial/401_CNN.py at master · MorvanZhou/PyTorch-Tutorial · GitHub
pytorch_tutorial/example1.py at master · soravux/pytorch_tutorial · GitHub
データの読み込み
import os import torch from torchvision import datasets, transforms from torch.autograd import Variable # load data data_root = os.path.expanduser('~/.torch/data/mnist') train_loader = torch.utils.data.DataLoader( datasets.MNIST(data_root, train=True, download=True, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.1307,), (0.3081,)) ])), shuffle=True, batch_size=32) test_loader = torch.utils.data.DataLoader( datasets.MNIST(data_root, train=False, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.1307,), (0.3081,)) ])), shuffle=True, batch_size=32) for i, (images, labels) in enumerate(train_loader): images = Variable(images) labels = Variable(labels) print i print images.data.shape print labels.data.shape
最後一行の結果
1874 (32L, 1L, 28L, 28L) (32L,)
データの個数
バッチサイズ、チャンネル、サイズ、サイズ
ラベルデータ
となっている。
学習:ケース1
pytorch.learning/mnist_conv.py at master · moskomule/pytorch.learning · GitHub
""" mnist classification which use nn module """ import os import torch import torch.nn as nn import torch.nn.functional as F import torch.optim as optim from torchvision import datasets, transforms from torch.autograd import Variable cuda = torch.cuda.is_available() # load data data_root = os.path.expanduser('~/.torch/data/mnist') train_loader = torch.utils.data.DataLoader( datasets.MNIST(data_root, train=True, download=True, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.1307,), (0.3081,)) ])), shuffle=True, batch_size=32) test_loader = torch.utils.data.DataLoader( datasets.MNIST(data_root, train=False, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.1307,), (0.3081,)) ])), shuffle=True, batch_size=32) class Net1(nn.Module): def __init__(self): super(Net1, self).__init__() self.conv1 = nn.Conv2d(in_channels=1, out_channels=10, kernel_size=5, stride=1) self.conv2 = nn.Conv2d(10, 20, kernel_size=5) self.dense1 = nn.Linear(in_features=320, out_features=50) self.dense2 = nn.Linear(50, 10) def forward(self, x): x = self.conv1(x) x = F.max_pool2d(x, kernel_size=2) x = F.relu(x) x = self.conv2(x) x = F.max_pool2d(x, 2) x = F.relu(x) print x x = x.view(-1, 320) x = self.dense1(x) x = F.relu(x) x = self.dense2(x) return F.log_softmax(x) # alternative way class Net2(nn.Module): def __init__(self): super(Net2, self).__init__() self.head = nn.Sequential( nn.Conv2d(in_channels=1, out_channels=10, kernel_size=5, stride=1), nn.MaxPool2d(kernel_size=2), nn.ReLU(), nn.Conv2d(10, 20, kernel_size=5), nn.MaxPool2d(kernel_size=2), nn.ReLU()) self.tail = nn.Sequential( nn.Linear(320, 50), nn.ReLU(), nn.Linear(50, 10)) def forward(self, x): x = self.head(x) x = x.view(-1, 320) x = self.tail(x) return F.log_softmax(x) def train(model, optimizer, epoch): model.train() for batch_idx, (data, target) in enumerate(train_loader): if cuda: data, target = data.cuda(), target.cuda() data, target = Variable(data), Variable(target) optimizer.zero_grad() # reset reset optimizer output = model(data) loss = F.nll_loss(output, target) # negative log likelihood loss loss.backward() # backprop optimizer.step() if batch_idx % 20 == 0: print('Train Epoch: {} [{}/{} ({:.0f}%)]\tLoss: {:.6f}'.format( epoch, batch_idx * len(data), len(train_loader.dataset), 100. * batch_idx / len(train_loader), loss.data[0])) def test(model): model.eval() test_loss = 0 correct = 0 for data, target in test_loader: if cuda: data, target = data.cuda(), target.cuda() data, target = Variable(data), Variable(target) output = model(data) test_loss += F.nll_loss(output, target).data[0] pred = output.data.max(1)[1] # get the index of the max log-probability correct += pred.eq(target.data).cpu().sum() test_loss /= len(test_loader) # loss function already averages over batch size print('\nTest set: Average loss: {:.4f}, Accuracy: {}/{} ({:.0f}%)\n'.format( test_loss, correct, len(test_loader.dataset), 100. * correct / len(test_loader.dataset))) return test_loss if __name__ == '__main__': model = Net1() epochs = 10 if cuda: model.cuda() optimizer = optim.Adam(model.parameters(), lr=5e-4) loss = [] for i in range(1, epochs + 1): train(model, optimizer, i) loss.append(test(model))
print x の値は [torch.FloatTensor of size 32x20x4x4]
全結合層320は20x4x4=320で計算できる。4は
(( 28 - ( 5 -1 )) / 2 - ( 5 - 1)) / 2
から計算できる。
学習:ケース2
GitHub - yunjey/pytorch-tutorial: PyTorch Tutorial for Deep Learning Researchers
import torch import torch.nn as nn import torchvision.datasets as dsets import torchvision.transforms as transforms from torch.autograd import Variable # Hyper Parameters num_epochs = 10 batch_size = 100 learning_rate = 0.001 # MNIST Dataset train_dataset = dsets.MNIST(root='./data/', train=True, transform=transforms.ToTensor(), download=True) test_dataset = dsets.MNIST(root='./data/', train=False, transform=transforms.ToTensor()) # Data Loader (Input Pipeline) train_loader = torch.utils.data.DataLoader(dataset=train_dataset, batch_size=batch_size, shuffle=True) test_loader = torch.utils.data.DataLoader(dataset=test_dataset, batch_size=batch_size, shuffle=False) # CNN Model (2 conv layer) class CNN(nn.Module): def __init__(self): super(CNN, self).__init__() self.layer1 = nn.Sequential( nn.Conv2d(1, 16, kernel_size=5, padding=2), nn.BatchNorm2d(16), nn.ReLU(), nn.MaxPool2d(2)) self.layer2 = nn.Sequential( nn.Conv2d(16, 32, kernel_size=5, padding=2), nn.BatchNorm2d(32), nn.ReLU(), nn.MaxPool2d(2)) self.fc = nn.Linear(7*7*32, 10) def forward(self, x): out = self.layer1(x) out = self.layer2(out) print out out = out.view(out.size(0), -1) out = self.fc(out) return out cnn = CNN() # Loss and Optimizer criterion = nn.CrossEntropyLoss() optimizer = torch.optim.Adam(cnn.parameters(), lr=learning_rate) # Train the Model for epoch in range(num_epochs): for i, (images, labels) in enumerate(train_loader): images = Variable(images) labels = Variable(labels) # Forward + Backward + Optimize optimizer.zero_grad() outputs = cnn(images) loss = criterion(outputs, labels) loss.backward() optimizer.step() if (i+1) % 100 == 0: print ('Epoch [%d/%d], Iter [%d/%d] Loss: %.4f' %(epoch+1, num_epochs, i+1, len(train_dataset)//batch_size, loss.data[0])) # Test the Model cnn.eval() # Change model to 'eval' mode (BN uses moving mean/var). correct = 0 total = 0 for images, labels in test_loader: images = Variable(images) outputs = cnn(images) _, predicted = torch.max(outputs.data, 1) total += labels.size(0) correct += (predicted == labels).sum() print('Test Accuracy of the model on the 10000 test images: %d %%' % (100 * correct / total)) # Save the Trained Model torch.save(cnn.state_dict(), 'cnn.pkl')
print x
[torch.FloatTensor of size 100x20x7x7]
out.size(0)=100
モデルの読み込み
model = models.resnet50(pretrained=True) # modelの保存 torch.save(model.state_dict(), 'weight.pth') model2 = models.resnet50() # パラメータの読み込み param = torch.load('weight.pth') model2.load_state_dict(param)
AweSome Pytorch List
Model Converter
Segmentation Sample
pspnet
あつい!試してみたい!! https://github.com/Lextal/pspnet-pytorch
機械学習のお勉強(次元削減)
教科書
特徴抽出
特徴抽出ではデータを新しい特徴空間に射影する
主成分分析(PCA)
線形判別分離(LDA)
カーネル主成分分析
などがある。
主成分分析による次元削減

d次元のデータセットを標準化する
標準化したデータセットの共分散行列を作成する
上位k個の固有ベクトルから射影行列Wを作成する
射影行列Wを使ってd次元の入力データセットXを変換し、新しいk次元の特徴部分空間を取得する
共分散行列の固有値
- 標準化
import pandas as pd df_wine = pd.read_csv('https://archive.ics.uci.edu/ml/' 'machine-learning-databases/wine/wine.data', header=None) df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash', 'Alcalinity of ash', 'Magnesium', 'Total phenols', 'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins', 'Color intensity', 'Hue', 'OD280/OD315 of diluted wines', 'Proline'] df_wine.head() if Version(sklearn_version) < '0.18': from sklearn.cross_validation import train_test_split else: from sklearn.model_selection import train_test_split X, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values X_train, X_test, y_train, y_test = \ train_test_split(X, y, test_size=0.3, random_state=0) from sklearn.preprocessing import StandardScaler sc = StandardScaler() X_train_std = sc.fit_transform(X_train) X_test_std = sc.transform(X_test)
import numpy as np cov_mat = np.cov(X_train_std.T) eigen_vals, eigen_vecs = np.linalg.eig(cov_mat) print('\nEigenvalues \n%s' % eigen_vals)
固有値の分散説明率
固有値 : lambda 分散説明率 : lambda_j / sum(lambda)
実装
tot = sum(eigen_vals) var_exp = [(i / tot) for i in sorted(eigen_vals, reverse=True)] cum_var_exp = np.cumsum(var_exp) import matplotlib.pyplot as plt plt.bar(range(1, 14), var_exp, alpha=0.5, align='center', label='individual explained variance') plt.step(range(1, 14), cum_var_exp, where='mid', label='cumulative explained variance') plt.ylabel('Explained variance ratio') plt.xlabel('Principal components') plt.legend(loc='best') plt.tight_layout() # plt.savefig('./figures/pca1.png', dpi=300) plt.show()
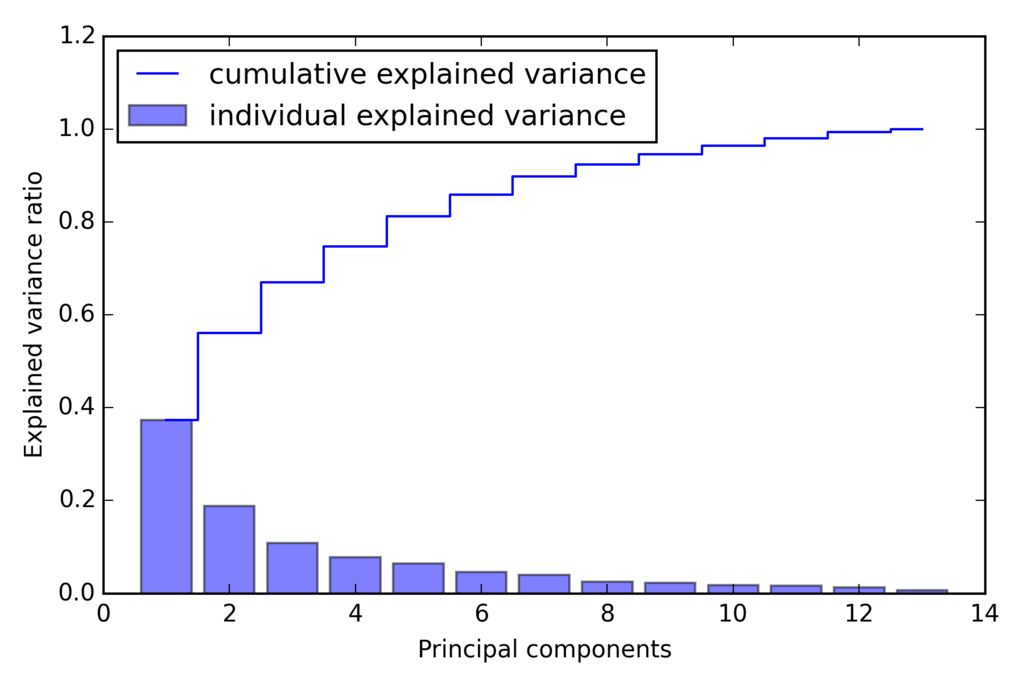
最初の2つの主成分で分散の60%近くになることがわかる。
ランダムフォレストのクラスラベルの重要度と似ているが、こちらはラベルを使わずデータ散らばり具合を利用して導出される。
特徴変換
4 . 固有値の大きいものから並べ替え
# Make a list of (eigenvalue, eigenvector) tuples eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:, i]) for i in range(len(eigen_vals))] # Sort the (eigenvalue, eigenvector) tuples from high to low eigen_pairs.sort(key=lambda k: k[0], reverse=True) # Note: I added the `key=lambda k: k[0]` in the sort call above # just like I used it further below in the LDA section. # This is to avoid problems if there are ties in the eigenvalue # arrays (i.e., the sorting algorithm will only regard the # first element of the tuples, now).
w = np.hstack((eigen_pairs[0][1][:, np.newaxis], eigen_pairs[1][1][:, np.newaxis])) print('Matrix W:\n', w)
この13x2次元の射影行列Wを用いて、2次元のサンプルベクトルx'を生成できる。
print X_train_std[0].dot(w) # array([ 2.59891628, 0.00484089])
6 . トレーニングデータセットを2次元の散布図としてプロット
X_train_pca = X_train_std.dot(w) colors = ['r', 'b', 'g'] markers = ['s', 'x', 'o'] for l, c, m in zip(np.unique(y_train), colors, markers): plt.scatter(X_train_pca[y_train == l, 0], X_train_pca[y_train == l, 1], c=c, label=l, marker=m) plt.xlabel('PC 1') plt.ylabel('PC 2') plt.legend(loc='lower left') plt.tight_layout() # plt.savefig('./figures/pca2.png', dpi=300) plt.show()

scikit-learnで主成分分析
from sklearn.decomposition import PCA pca = PCA() X_train_pca = pca.fit_transform(X_train_std) pca.explained_variance_ratio_
上位2つに対してPCA
pca = PCA(n_components=2)
X_train_pca = pca.fit_transform(X_train_std)
X_test_pca = pca.transform(X_test_std)
ロジスティック回帰実施
from sklearn.linear_model import LogisticRegression lr = LogisticRegression() lr = lr.fit(X_train_pca, y_train)
train結果

test結果

この結果からロジスティック回帰の性能が良いことがわかる
また、すべての主成分の分散説明率を知りたい場合、n_components=Noneを設定して初期化する。
pca = PCA(n_components=None) X_train_pca = pca.fit_transform(X_train_std) pca.explained_variance_ratio_
線形判別分析によるデータ圧縮
計算効率を高め、正規化されていないモデルで次元の呪いによる過学習を抑制することができる
クラスの分離を最適化する特徴部分空間を見つけ出す
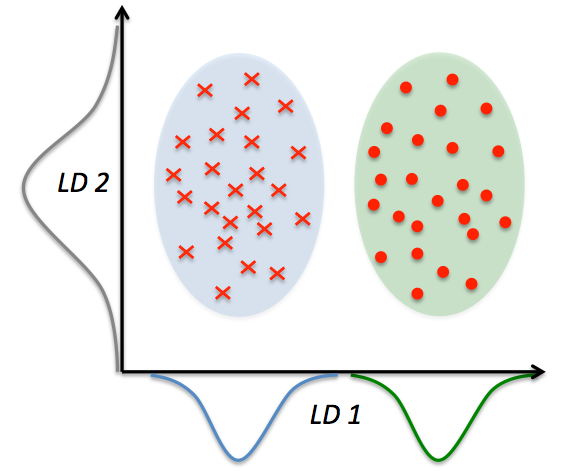
d次元のデータセットを標準化する
クラス毎にd次元の平均ベクトルを計算する
クラス間変動行列Sbとクラス内変動行列Swを生成する
dxk次元の変換行列Wを生成するためにもっとも大きいk個の固有値に対応するk個の固有ベクトルを選択する。(固有ベクトルは行列の列)
変換行列Wを使ってサンプルを新しい特徴部分空間へ射影する
変動行列を計算
2 . 平均ベクトル
np.set_printoptions(precision=4) mean_vecs = [] for label in range(1, 4): mean_vecs.append(np.mean(X_train_std[y_train == label], axis=0)) print('MV %s: %s\n' % (label, mean_vecs[label - 1]))
3 . クラス内変動行列
d = 13 # number of features S_W = np.zeros((d, d)) for label, mv in zip(range(1, 4), mean_vecs): class_scatter = np.zeros((d, d)) # scatter matrix for each class for row in X_train_std[y_train == label]: row, mv = row.reshape(d, 1), mv.reshape(d, 1) # make column vectors class_scatter += (row - mv).dot((row - mv).T) S_W += class_scatter # sum class scatter matrices print('Within-class scatter matrix: %sx%s' % (S_W.shape[0], S_W.shape[1])) # Within-class scatter matrix: 13x13
上記を行うにはクラスラベルが一様に分布している必要がある
しかし、
print('Class label distribution: %s' % np.bincount(y_train)[1:]) # Class label distribution: [40 49 35]
満たしていないので、スケーリングが必要。スケーリング、サンプル数で割る、操作を行うと共分散行列と同じであることがわかる。
よって以下で表すことができる
d = 13 # number of features S_W = np.zeros((d, d)) for label, mv in zip(range(1, 4), mean_vecs): class_scatter = np.cov(X_train_std[y_train == label].T) S_W += class_scatter print('Scaled within-class scatter matrix: %sx%s' % (S_W.shape[0], S_W.shape[1]))
3 . クラス間変動行列
mean_overall = np.mean(X_train_std, axis=0) d = 13 # number of features S_B = np.zeros((d, d)) for i, mean_vec in enumerate(mean_vecs): n = X_train[y_train == i + 1, :].shape[0] mean_vec = mean_vec.reshape(d, 1) # make column vector mean_overall = mean_overall.reshape(d, 1) # make column vector S_B += n * (mean_vec - mean_overall).dot((mean_vec - mean_overall).T) print('Between-class scatter matrix: %sx%s' % (S_B.shape[0], S_B.shape[1])) # Between-class scatter matrix: 13x13
新しい特徴部分空間の線形判別を選択
4 . 固有値問題を解く
eigen_vals, eigen_vecs = np.linalg.eig(np.linalg.inv(S_W).dot(S_B)) # Make a list of (eigenvalue, eigenvector) tuples eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:, i]) for i in range(len(eigen_vals))] # Sort the (eigenvalue, eigenvector) tuples from high to low eigen_pairs = sorted(eigen_pairs, key=lambda k: k[0], reverse=True) # Visually confirm that the list is correctly sorted by decreasing eigenvalues print('Eigenvalues in decreasing order:\n') for eigen_val in eigen_pairs: print(eigen_val[0])
結果
Eigenvalues in decreasing order: 452.721581245 156.43636122 3.59696053827e-14 3.50722067103e-14 3.50722067103e-14 2.84217094304e-14 2.46524103582e-14 2.46524103582e-14 2.18345154287e-14 2.18345154287e-14 1.69066127548e-14 1.39492602566e-15 1.39492602566e-15
上記からわかる通り、LDAでの線形判別の個数は最大で(クラス数-1)個となる。
TODO:なぜLDAでの線形判別の個数は最大で(クラス数-1)個となるのか調べる。。。わかる方教えてください笑
線形判別のプロット
tot = sum(eigen_vals.real) discr = [(i / tot) for i in sorted(eigen_vals.real, reverse=True)] cum_discr = np.cumsum(discr) plt.bar(range(1, 14), discr, alpha=0.5, align='center', label='individual "discriminability"') plt.step(range(1, 14), cum_discr, where='mid', label='cumulative "discriminability"') plt.ylabel('"discriminability" ratio') plt.xlabel('Linear Discriminants') plt.ylim([-0.1, 1.1]) plt.legend(loc='best') plt.tight_layout() # plt.savefig('./figures/lda1.png', dpi=300) plt.show()
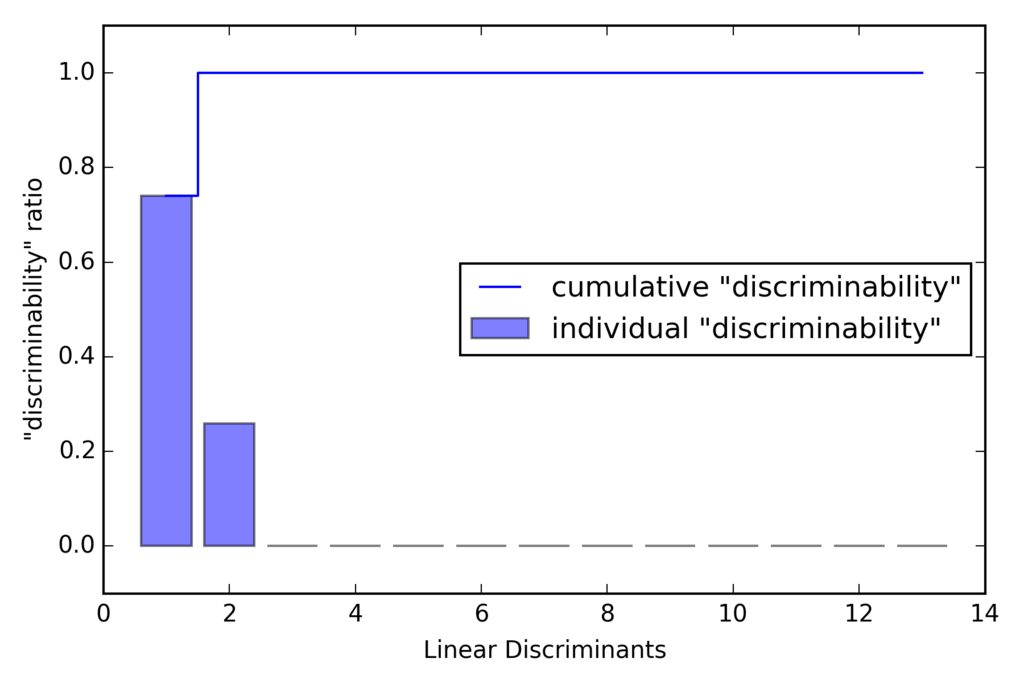
5 . 変換行列の作成
w = np.hstack((eigen_pairs[0][1][:, np.newaxis].real, eigen_pairs[1][1][:, np.newaxis].real)) print('Matrix W:\n', w)
結果
w = np.hstack((eigen_pairs[0][1][:, np.newaxis].real, eigen_pairs[1][1][:, np.newaxis].real)) print('Matrix W:\n', w) w = np.hstack((eigen_pairs[0][1][:, np.newaxis].real, eigen_pairs[1][1][:, np.newaxis].real)) print('Matrix W:\n', w) Matrix W: [[-0.0662 -0.3797] [ 0.0386 -0.2206] [-0.0217 -0.3816] [ 0.184 0.3018] [-0.0034 0.0141] [ 0.2326 0.0234] [-0.7747 0.1869] [-0.0811 0.0696] [ 0.0875 0.1796] [ 0.185 -0.284 ] [-0.066 0.2349] [-0.3805 0.073 ] [-0.3285 -0.5971]]
新しい特徴空間にサンプルを射影
- トレーニングデータセットの変換
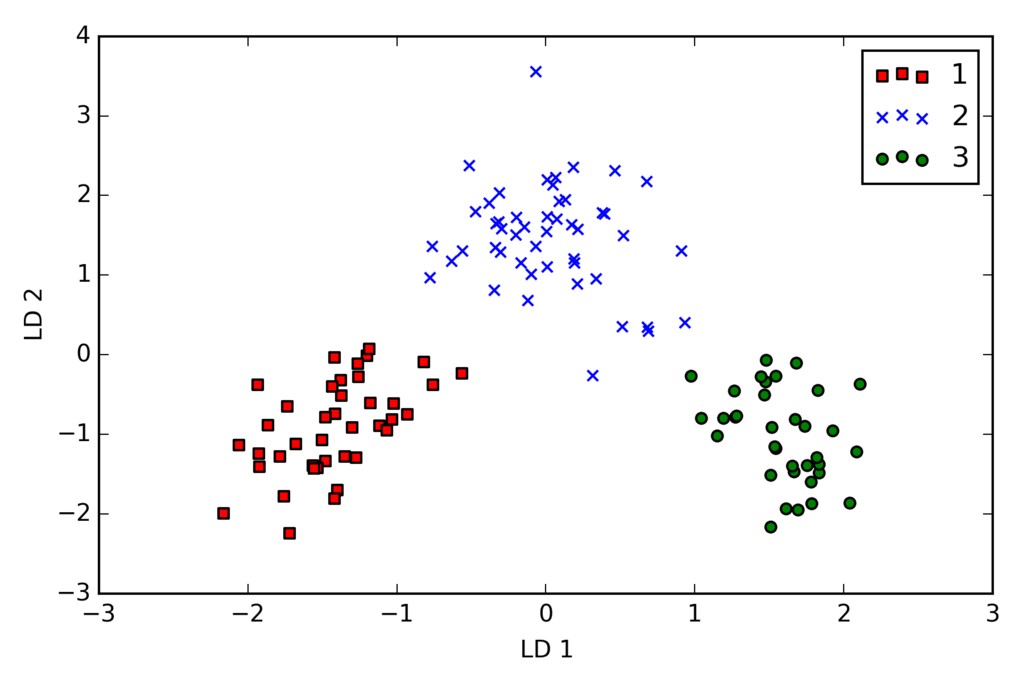
X_train_lda = X_train_std.dot(w) colors = ['r', 'b', 'g'] markers = ['s', 'x', 'o'] for l, c, m in zip(np.unique(y_train), colors, markers): plt.scatter(X_train_lda[y_train == l, 0] * (-1), X_train_lda[y_train == l, 1] * (-1), c=c, label=l, marker=m) plt.xlabel('LD 1') plt.ylabel('LD 2') plt.legend(loc='lower right') plt.tight_layout() # plt.savefig('./figures/lda2.png', dpi=300) plt.show()
scikit-learnによるLDA
if Version(sklearn_version) < '0.18': from sklearn.lda import LDA else: from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA lda = LDA(n_components=2) X_train_lda = lda.fit_transform(X_train_std, y_train)
ロジスティック回帰でトレーニング
from sklearn.linear_model import LogisticRegression lr = LogisticRegression() lr = lr.fit(X_train_lda, y_train) plot_decision_regions(X_train_lda, y_train, classifier=lr) plt.xlabel('LD 1') plt.ylabel('LD 2') plt.legend(loc='lower left') plt.tight_layout() # plt.savefig('./images/lda3.png', dpi=300) plt.show()

テストデータ分類
X_test_lda = lda.transform(X_test_std) plot_decision_regions(X_test_lda, y_test, classifier=lr) plt.xlabel('LD 1') plt.ylabel('LD 2') plt.legend(loc='lower left') plt.tight_layout() # plt.savefig('./images/lda4.png', dpi=300) plt.show()

2次元の特徴部分空間だけを用いてすべて正しく分類できている
カーネル主成分分析を使った非線形写像
線形に分離できないデータを変換し、線形分類器に適した新しい低次元の部分空間へ射影する
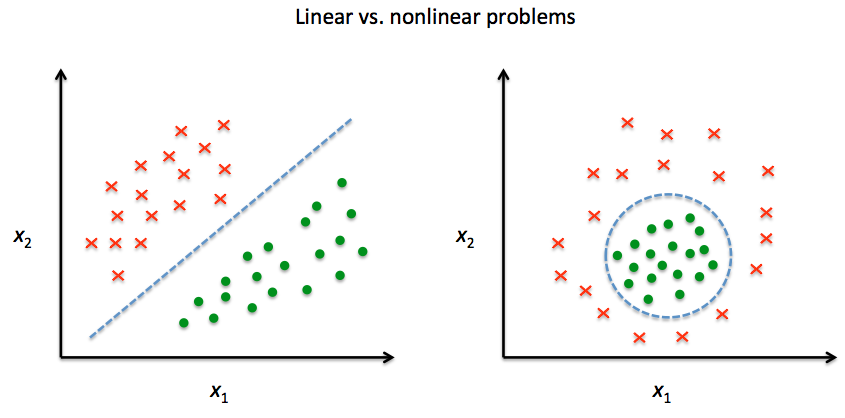
カーネル関数とカーネルトリック
双曲線正接カーネル:サポートベクターマシンとは[カーネル法による非線形サポートベクターマシン] - verum ipsum factum
動径基底関数、ガウスカーネル:放射基底関数(Radial basis function, RBF) - 大人になってからの再学習
カーネル行列を計算 : K(xi, xj) = exp(-gamma * ||x_i - x_j ||**2)
カーネル行列の中心化
実装
from scipy.spatial.distance import pdist, squareform from scipy import exp from numpy.linalg import eigh import numpy as np def rbf_kernel_pca(X, gamma, n_components): """ RBF kernel PCA implementation. Parameters ------------ X: {NumPy ndarray}, shape = [n_samples, n_features] gamma: float Tuning parameter of the RBF kernel n_components: int Number of principal components to return Returns ------------ X_pc: {NumPy ndarray}, shape = [n_samples, k_features] Projected dataset """ # Calculate pairwise squared Euclidean distances # in the MxN dimensional dataset. sq_dists = pdist(X, 'sqeuclidean') # Convert pairwise distances into a square matrix. mat_sq_dists = squareform(sq_dists) # Compute the symmetric kernel matrix. K = exp(-gamma * mat_sq_dists) # Center the kernel matrix. N = K.shape[0] one_n = np.ones((N, N)) / N K = K - one_n.dot(K) - K.dot(one_n) + one_n.dot(K).dot(one_n) # Obtaining eigenpairs from the centered kernel matrix # numpy.linalg.eigh returns them in sorted order eigvals, eigvecs = eigh(K) # Collect the top k eigenvectors (projected samples) X_pc = np.column_stack((eigvecs[:, -i] for i in range(1, n_components + 1))) return X_pc
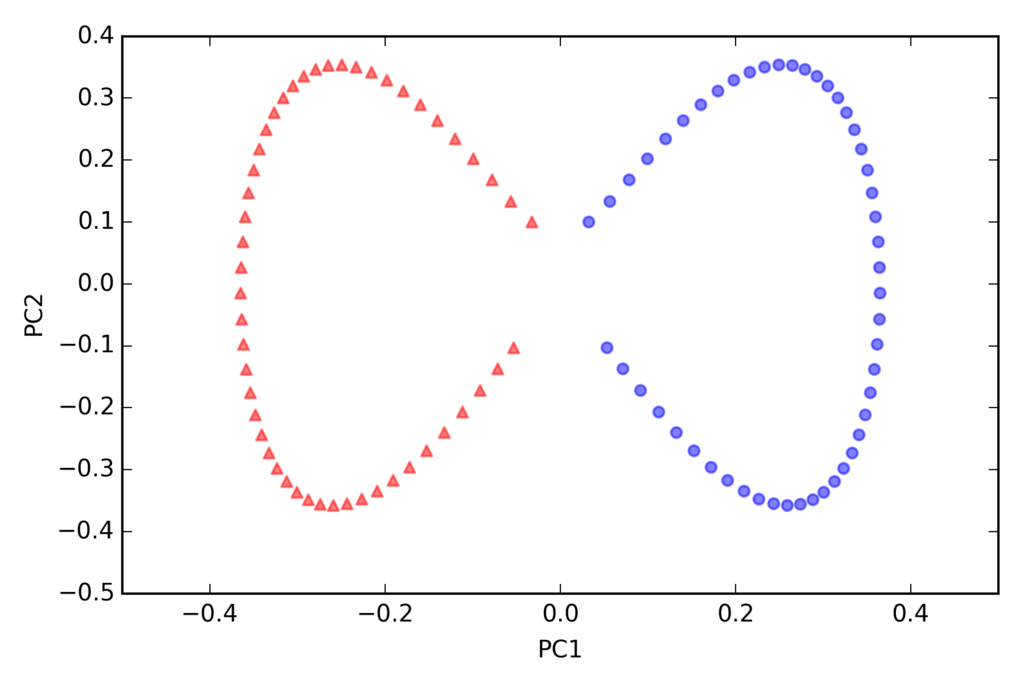
半月型の分離
import matplotlib.pyplot as plt from sklearn.datasets import make_moons X, y = make_moons(n_samples=100, random_state=123) plt.scatter(X[y == 0, 0], X[y == 0, 1], color='red', marker='^', alpha=0.5) plt.scatter(X[y == 1, 0], X[y == 1, 1], color='blue', marker='o', alpha=0.5) plt.tight_layout() # plt.savefig('./figures/half_moon_1.png', dpi=300) plt.show()

標準のPCA
from sklearn.decomposition import PCA from sklearn.preprocessing import StandardScaler scikit_pca = PCA(n_components=2) X_spca = scikit_pca.fit_transform(X) fig, ax = plt.subplots(nrows=1, ncols=2, figsize=(7, 3)) ax[0].scatter(X_spca[y == 0, 0], X_spca[y == 0, 1], color='red', marker='^', alpha=0.5) ax[0].scatter(X_spca[y == 1, 0], X_spca[y == 1, 1], color='blue', marker='o', alpha=0.5) ax[1].scatter(X_spca[y == 0, 0], np.zeros((50, 1)) + 0.02, color='red', marker='^', alpha=0.5) ax[1].scatter(X_spca[y == 1, 0], np.zeros((50, 1)) - 0.02, color='blue', marker='o', alpha=0.5) ax[0].set_xlabel('PC1') ax[0].set_ylabel('PC2') ax[1].set_ylim([-1, 1]) ax[1].set_yticks([]) ax[1].set_xlabel('PC1') plt.tight_layout() # plt.savefig('./figures/half_moon_2.png', dpi=300) plt.show()
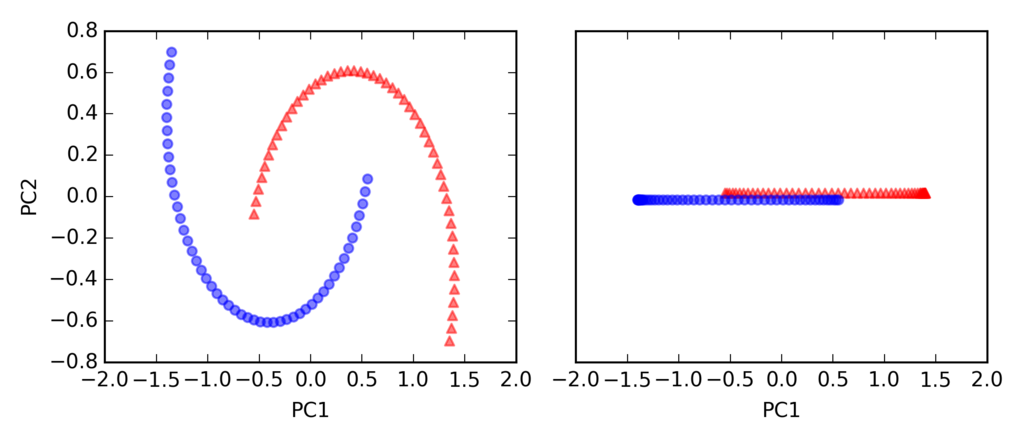
カーネルPCA
from matplotlib.ticker import FormatStrFormatter X_kpca = rbf_kernel_pca(X, gamma=15, n_components=2) fig, ax = plt.subplots(nrows=1,ncols=2, figsize=(7,3)) ax[0].scatter(X_kpca[y==0, 0], X_kpca[y==0, 1], color='red', marker='^', alpha=0.5) ax[0].scatter(X_kpca[y==1, 0], X_kpca[y==1, 1], color='blue', marker='o', alpha=0.5) ax[1].scatter(X_kpca[y==0, 0], np.zeros((50,1))+0.02, color='red', marker='^', alpha=0.5) ax[1].scatter(X_kpca[y==1, 0], np.zeros((50,1))-0.02, color='blue', marker='o', alpha=0.5) ax[0].set_xlabel('PC1') ax[0].set_ylabel('PC2') ax[1].set_ylim([-1, 1]) ax[1].set_yticks([]) ax[1].set_xlabel('PC1') ax[0].xaxis.set_major_formatter(FormatStrFormatter('%0.1f')) ax[1].xaxis.set_major_formatter(FormatStrFormatter('%0.1f')) plt.tight_layout() # plt.savefig('./figures/half_moon_3.png', dpi=300) plt.show()

rbf_kernel_pca(X, gamma=15, n_components=2)のgammaはチューニングパラメータで適切な値を設定する必要がある
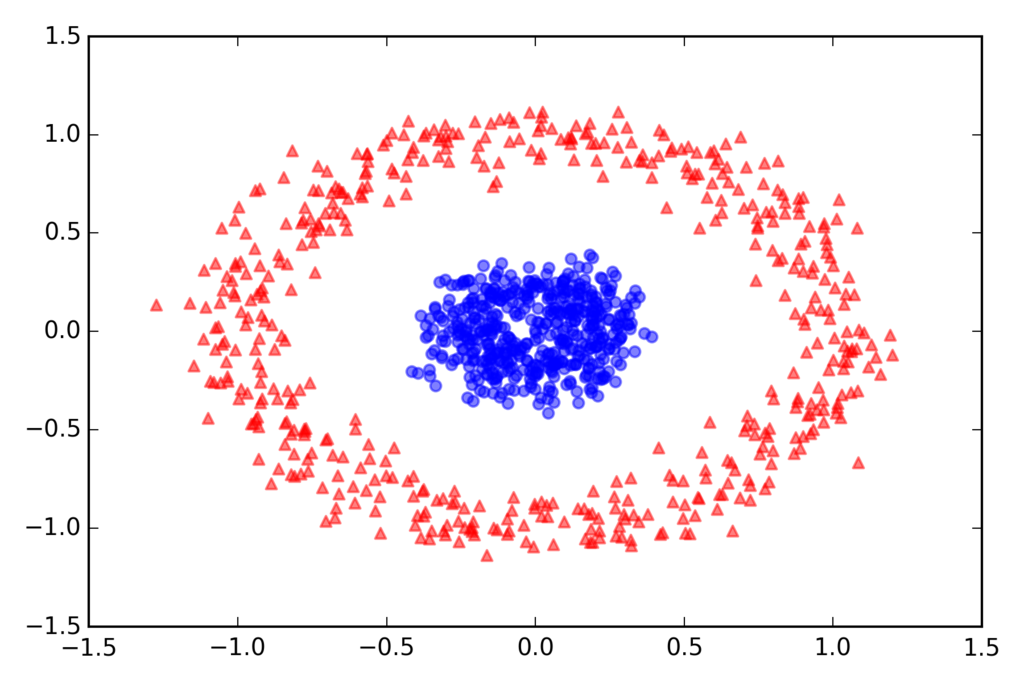
同心円の分離
from sklearn.datasets import make_circles X, y = make_circles(n_samples=1000, random_state=123, noise=0.1, factor=0.2) plt.scatter(X[y == 0, 0], X[y == 0, 1], color='red', marker='^', alpha=0.5) plt.scatter(X[y == 1, 0], X[y == 1, 1], color='blue', marker='o', alpha=0.5) plt.tight_layout() # plt.savefig('./figures/circles_1.png', dpi=300) plt.show()
標準のPCA
scikit_pca = PCA(n_components=2) X_spca = scikit_pca.fit_transform(X) fig, ax = plt.subplots(nrows=1, ncols=2, figsize=(7, 3)) ax[0].scatter(X_spca[y == 0, 0], X_spca[y == 0, 1], color='red', marker='^', alpha=0.5) ax[0].scatter(X_spca[y == 1, 0], X_spca[y == 1, 1], color='blue', marker='o', alpha=0.5) ax[1].scatter(X_spca[y == 0, 0], np.zeros((500, 1)) + 0.02, color='red', marker='^', alpha=0.5) ax[1].scatter(X_spca[y == 1, 0], np.zeros((500, 1)) - 0.02, color='blue', marker='o', alpha=0.5) ax[0].set_xlabel('PC1') ax[0].set_ylabel('PC2') ax[1].set_ylim([-1, 1]) ax[1].set_yticks([]) ax[1].set_xlabel('PC1') plt.tight_layout() # plt.savefig('./figures/circles_2.png', dpi=300) plt.show()
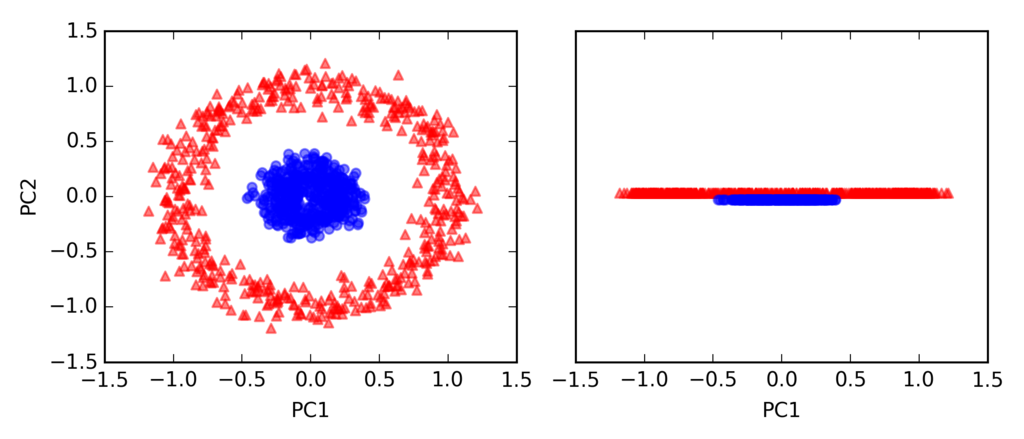
カーネルPCA
X_kpca = rbf_kernel_pca(X, gamma=15, n_components=2) fig, ax = plt.subplots(nrows=1, ncols=2, figsize=(7, 3)) ax[0].scatter(X_kpca[y == 0, 0], X_kpca[y == 0, 1], color='red', marker='^', alpha=0.5) ax[0].scatter(X_kpca[y == 1, 0], X_kpca[y == 1, 1], color='blue', marker='o', alpha=0.5) ax[1].scatter(X_kpca[y == 0, 0], np.zeros((500, 1)) + 0.02, color='red', marker='^', alpha=0.5) ax[1].scatter(X_kpca[y == 1, 0], np.zeros((500, 1)) - 0.02, color='blue', marker='o', alpha=0.5) ax[0].set_xlabel('PC1') ax[0].set_ylabel('PC2') ax[1].set_ylim([-1, 1]) ax[1].set_yticks([]) ax[1].set_xlabel('PC1') plt.tight_layout() # plt.savefig('./figures/circles_3.png', dpi=300) plt.show()

新しいデータ点の射影
from scipy.spatial.distance import pdist, squareform from scipy import exp from scipy.linalg import eigh import numpy as np def rbf_kernel_pca(X, gamma, n_components): """ RBF kernel PCA implementation. Parameters ------------ X: {NumPy ndarray}, shape = [n_samples, n_features] gamma: float Tuning parameter of the RBF kernel n_components: int Number of principal components to return Returns ------------ X_pc: {NumPy ndarray}, shape = [n_samples, k_features] Projected dataset lambdas: list Eigenvalues """ # Calculate pairwise squared Euclidean distances # in the MxN dimensional dataset. sq_dists = pdist(X, 'sqeuclidean') # Convert pairwise distances into a square matrix. mat_sq_dists = squareform(sq_dists) # Compute the symmetric kernel matrix. K = exp(-gamma * mat_sq_dists) # Center the kernel matrix. N = K.shape[0] one_n = np.ones((N, N)) / N K = K - one_n.dot(K) - K.dot(one_n) + one_n.dot(K).dot(one_n) # Obtaining eigenpairs from the centered kernel matrix # numpy.eigh returns them in sorted order eigvals, eigvecs = eigh(K) # Collect the top k eigenvectors (projected samples) alphas = np.column_stack((eigvecs[:, -i] for i in range(1, n_components + 1))) # Collect the corresponding eigenvalues lambdas = [eigvals[-i] for i in range(1, n_components + 1)] return alphas, lambdas
一次元の部分空間に射影
X, y = make_moons(n_samples=100, random_state=123) alphas, lambdas = rbf_kernel_pca(X, gamma=15, n_components=1) x_new = X[-1] x_new x_proj = alphas[-1] # original projection x_proj def project_x(x_new, X, gamma, alphas, lambdas): pair_dist = np.array([np.sum((x_new - row)**2) for row in X]) k = np.exp(-gamma * pair_dist) return k.dot(alphas / lambdas) # projection of the "new" datapoint x_reproj = project_x(x_new, X, gamma=15, alphas=alphas, lambdas=lambdas) x_reproj plt.scatter(alphas[y == 0, 0], np.zeros((50)), color='red', marker='^', alpha=0.5) plt.scatter(alphas[y == 1, 0], np.zeros((50)), color='blue', marker='o', [f:id:robonchu:20171016215610p:plain]alpha=0.5) plt.scatter(x_proj, 0, color='black', label='original projection of point X[25]', marker='^', s=100) plt.scatter(x_reproj, 0, color='green', label='remapped point X[25]', marker='x', s=500) plt.legend(scatterpoints=1) plt.tight_layout() # plt.savefig('./figures/reproject.png', dpi=300) plt.show()
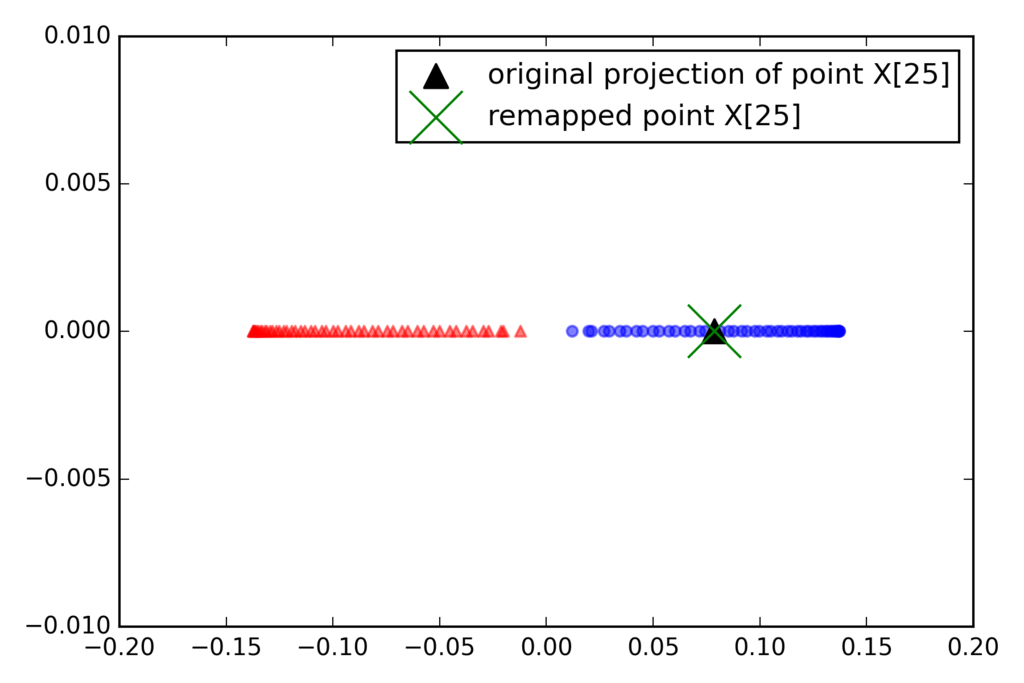
scikit-learnのカーネルPCA
from sklearn.decomposition import KernelPCA X, y = make_moons(n_samples=100, random_state=123) scikit_kpca = KernelPCA(n_components=2, kernel='rbf', gamma=15) X_skernpca = scikit_kpca.fit_transform(X) plt.scatter(X_skernpca[y == 0, 0], X_skernpca[y == 0, 1], color='red', marker='^', alpha=0.5) plt.scatter(X_skernpca[y == 1, 0], X_skernpca[y == 1, 1], color='blue', marker='o', alpha=0.5) plt.xlabel('PC1') plt.ylabel('PC2') plt.tight_layout() # plt.savefig('./figures/scikit_kpca.png', dpi=300) plt.show()
その他の手法
機械学習のお勉強(データの前処理)①
教科書
データセットの欠損値の削除と補完
pandasを使ってデータを読み込むと欠損場所にNanが入る。 欠損値のカウントを行うこともでき、numpyにはvaluesによってアクセスできる。
import pandas as pd from io import StringIO csv_data = '''A,B,C,D 1,2,3,4 5,6,,8 10,11,12,''' csv_data = unicode(csv_data) df = pd.read_csv(StringIO(csv_data)) print df print df.values print df.isnull().sum()
削除
print df.dropna() print df.dropna(axis=1)
補完
from sklearn.preprocessing import Imputer imr = Imputer(missing_values='NaN', strategy='mean',axis=0) imr = imr.fit(df) imputed_data = imr.transform(df.values) imputed_data
機械学習のアルゴリズムに合わせたカテゴリデータの整形
順序特徴量のマッピング
df = pd.DataFrame([
['green', 'M',10.1,'class1'],
['red','L',13.5,'class2'],
['blue','XL',15.3,'class1']])
df.columns = ['color' , 'size', 'price', 'classlabel']
size_mapping ={'XL':3, 'L':2,'M':1}
df['size'] = df['size'].map(size_mapping)
print df
クラスラベルのエンコーディング
import numpy as np class_mapping = {label:idx for idx,label in enumerate(np.unique(df['classlabel']))} print class_mapping df['classlabel'] = df['classlabel'].map(class_mapping) print df inv_class_mapping = {v: k for k,v in class_mapping.items()} df['classlabel'] = df['classlabel'].map(inv_class_mapping) print df
Label Encoder
from sklearn.preprocessing import LabelEncoder class_le = LabelEncoder() y = class_le.fit_transform(df['classlabel'].values) print y print class_le.inverse_transform(y)
one-hotエンコーディング
X = df[['color', 'size', 'price']].values print X color_le = LabelEncoder() X[:, 0] = color_le.fit_transform(X[:, 0]) print X from sklearn.preprocessing import OneHotEncoder ohe = OneHotEncoder(categorical_features=[0]) print ohe.fit_transform(X).toarray() print pd.get_dummies(df[['price', 'color', 'size']])
データセットの分割
df_wine = pd.read_csv('https://raw.githubusercontent.com/rasbt/python-machine-learning-book/master/code/datasets/wine/wine.data', header=None) df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash', 'Alcalinity of ash', 'Magnesium', 'Total phenols', 'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins', 'Color intensity', 'Hue', 'OD280/OD315 of diluted wines', 'Proline'] df_wine.head() if Version(sklearn_version) < '0.18': from sklearn.cross_validation import train_test_split else: from sklearn.model_selection import train_test_split X, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values X_train, X_test, y_train, y_test = \ train_test_split(X, y, test_size=0.3, random_state=0)
標準化・正規化
正規化 0 ~ 1
from sklearn.preprocessing import MinMaxScaler mms = MinMaxScaler() X_train_norm = mms.fit_transform(X_train) # fit and trans X_test_norm = mms.transform(X_test) # trans only
標準化 平均:0 , 標準偏差:1
from sklearn.preprocessing import StandardScaler stdsc = StandardScaler() X_train_std = stdsc.fit_transform(X_train) X_test_std = stdsc.transform(X_test)
example
ex = pd.DataFrame([0, 1, 2, 3, 4, 5]) # standardize ex[1] = (ex[0] - ex[0].mean()) / ex[0].std(ddof=0) # Please note that pandas uses ddof=1 (sample standard deviation) # by default, whereas NumPy's std method and the StandardScaler # uses ddof=0 (population standard deviation) # normalize ex[2] = (ex[0] - ex[0].min()) / (ex[0].max() - ex[0].min()) ex.columns = ['input', 'standardized', 'normalized'] print ex
モデルの構築に適した特徴量の選択
汎化誤差を減らすための方法 1. 多くのデータを集める
正規化を通じて複雑さを減らす
パラメータの少ないシンプルなモデルを用いる
データの次元数を減らす
特徴量の選択:L1正規化
L2
L1

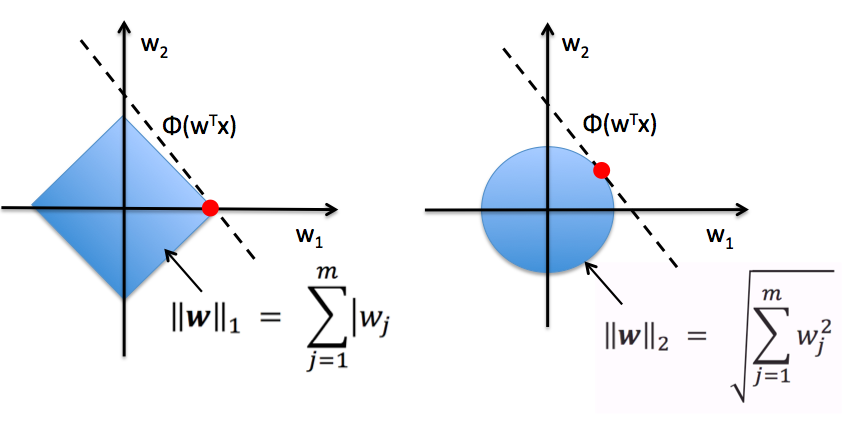
L1正規化は重みが軸上に乗ることが多く、疎な解が求まりやすく、特徴量の選択に有用。
無関係の特徴量が多い高次元のデータセットなどでの学習の場合、汎化性能の良い結果が得られる。
from sklearn.linear_model import LogisticRegression lr = LogisticRegression(penalty='l1', C=0.1) lr.fit(X_train_std, y_train) print('Training accuracy:', lr.score(X_train_std, y_train)) print('Test accuracy:', lr.score(X_test_std, y_test)) print lr.coef_
正規化の強さに対する特徴量の重み
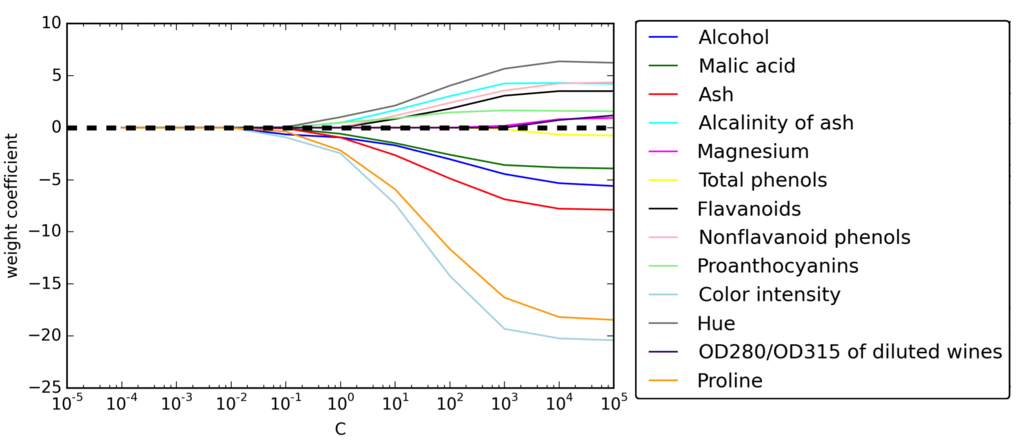
L1正規化のペナルティを増やすと特徴量がすべて0になる
次元削減(特徴選択・特徴抽出):特徴選択
フィルタ法、ラッパー法、埋め込み法の3つに大別される。
フィルタ法:情報利得やGini係数などを用いて、学習を伴わずに特徴量の重要性を測定して、有効な特徴量を選択する手法
ラッパー法:学習を行いながら重要な特徴量を選択する手法
埋め込み法:学習アルゴリズムに特徴量の選択も埋め込む。L1正規化、LASSOなどが該当。
逐次特徴選択アルゴリズム
目的
問題に最も関連がある特徴量の部分集合を自動的に選択する
無関係の特徴量やノイズを取り除くことで、モデルの汎化誤差を削減する
逐次後退選択
d次元の特徴空間をk次元の特徴部分空間に削除する
アルゴリズムをk=dで初期化
Jの評価を最大化する特徴量x^を決定する。x^ = argmax J(Xk - x) 。 x ∈Xk 。
特徴量の集合から特徴量x^を削除する
kが目的とする特徴量の個数に等しくなれば終了。そうでなければstep2へ。
実装
from sklearn.base import clone from itertools import combinations import numpy as np from sklearn.metrics import accuracy_score if Version(sklearn_version) < '0.18': from sklearn.cross_validation import train_test_split else: from sklearn.model_selection import train_test_split class SBS(): def __init__(self, estimator, k_features, scoring=accuracy_score, test_size=0.25, random_state=1): self.scoring = scoring self.estimator = clone(estimator) self.k_features = k_features self.test_size = test_size self.random_state = random_state def fit(self, X, y): X_train, X_test, y_train, y_test = \ train_test_split(X, y, test_size=self.test_size, random_state=self.random_state) dim = X_train.shape[1] self.indices_ = tuple(range(dim)) self.subsets_ = [self.indices_] score = self._calc_score(X_train, y_train, X_test, y_test, self.indices_) self.scores_ = [score] while dim > self.k_features: scores = [] subsets = [] for p in combinations(self.indices_, r=dim - 1): score = self._calc_score(X_train, y_train, X_test, y_test, p) scores.append(score) subsets.append(p) best = np.argmax(scores) self.indices_ = subsets[best] self.subsets_.append(self.indices_) dim -= 1 self.scores_.append(scores[best]) self.k_score_ = self.scores_[-1] return self def transform(self, X): return X[:, self.indices_] def _calc_score(self, X_train, y_train, X_test, y_test, indices): self.estimator.fit(X_train[:, indices], y_train) y_pred = self.estimator.predict(X_test[:, indices]) score = self.scoring(y_test, y_pred) return score
KNNで実装
import matplotlib.pyplot as plt from sklearn.neighbors import KNeighborsClassifier knn = KNeighborsClassifier(n_neighbors=2) # selecting features sbs = SBS(knn, k_features=1) sbs.fit(X_train_std, y_train) # plotting performance of feature subsets k_feat = [len(k) for k in sbs.subsets_] plt.plot(k_feat, sbs.scores_, marker='o') plt.ylim([0.7, 1.1]) plt.ylabel('Accuracy') plt.xlabel('Number of features') plt.grid() plt.tight_layout() # plt.savefig('./sbs.png', dpi=300) plt.show()
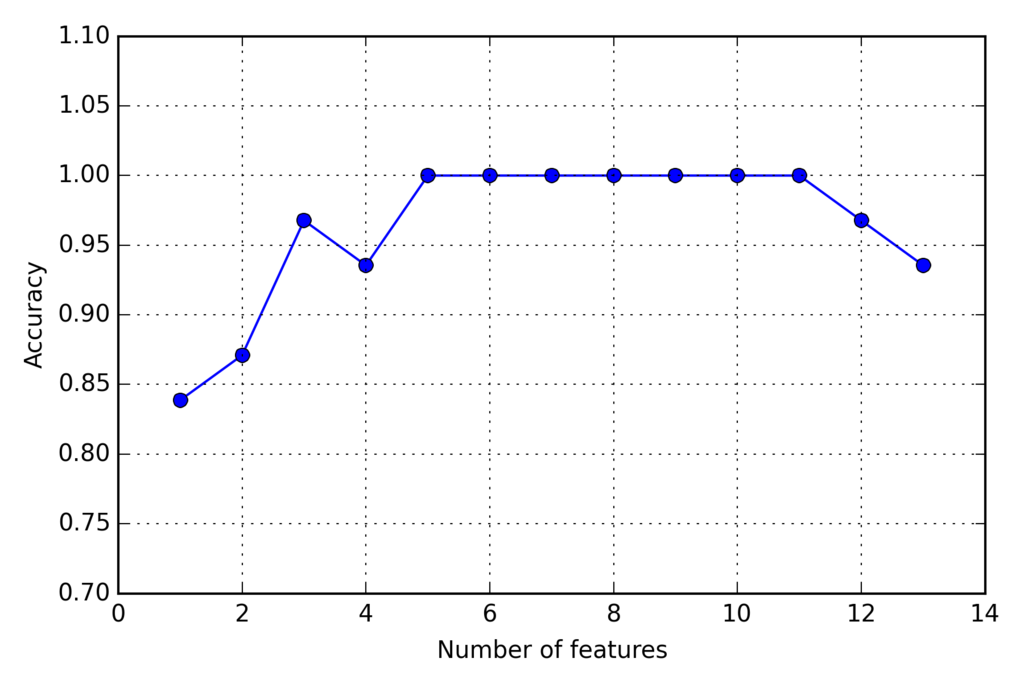 次元削減されたk=5で分類器が100%の正解率を達成している
次元削減されたk=5で分類器が100%の正解率を達成している
選ばれた特徴量は
k5 = list(sbs.subsets_[8]) print(df_wine.columns[1:][k5])
以下の5つ
(['Alcohol', 'Malic acid', 'Alcalinity of ash', 'Hue', 'Proline'], dtype='object')
次元削減前
knn.fit(X_train_std, y_train) print('Training accuracy:', knn.score(X_train_std, y_train)) print('Test accuracy:', knn.score(X_test_std, y_test))
結果
Training accuracy: 0.983870967742 Test accuracy: 0.944444444444
テストでの結果減。若干の過学習の傾向。
次元削減後
knn.fit(X_train_std[:, k5], y_train) print('Training accuracy:', knn.score(X_train_std[:, k5], y_train)) print('Test accuracy:', knn.score(X_test_std[:, k5], y_test))
結果
Training accuracy: 0.959677419355 Test accuracy: 0.962962962963
テストでの結果向上。過学習が軽減。
ランダムフォレストで特徴量の重要度測定
線形分離可能かどうかについて、前提を設けなくても、フォレスト内のすべての決定木から計算された不純度の平均的な減少量として特徴量の重要度を測定できる。
Winwデータセットの13個の特徴量の重要度に基づいてランク付け
from sklearn.ensemble import RandomForestClassifier feat_labels = df_wine.columns[1:] forest = RandomForestClassifier(n_estimators=10000, random_state=0, n_jobs=-1) forest.fit(X_train, y_train) importances = forest.feature_importances_ indices = np.argsort(importances)[::-1] for f in range(X_train.shape[1]): print("%2d) %-*s %f" % (f + 1, 30, feat_labels[indices[f]], importances[indices[f]])) plt.title('Feature Importances') plt.bar(range(X_train.shape[1]), importances[indices], color='lightblue', align='center') plt.xticks(range(X_train.shape[1]), feat_labels[indices], rotation=90) plt.xlim([-1, X_train.shape[1]]) plt.tight_layout() #plt.savefig('./random_forest.png', dpi=300) plt.show()
重要度の総和が0になるように正規化されている。

閾値で重要度の高い特徴量の抽出
if Version(sklearn_version) < '0.18': X_selected = forest.transform(X_train, threshold=0.15) else: from sklearn.feature_selection import SelectFromModel sfm = SelectFromModel(forest, threshold=0.15, prefit=True) X_selected = sfm.transform(X_train) print X_selected.shape for f in range(X_selected.shape[1]): print("%2d) %-*s %f" % (f + 1, 30, feat_labels[indices[f]], importances[indices[f]]))
重要度の高い3つが選ばれる
1) Color intensity 0.182483 2) Proline 0.158610 3) Flavanoids 0.150948
他の特徴量選択の手法
pythonでparrot mamboを飛ばしてみる
pymamboのセッテイング
https://github.com/amymcgovern/pymambo 👈 最高のライブラリ
の手順を実施
bluepyのインストール
sudo pip install bluepy
bluepyのissue
/usr/local/lib/python3.5/dist-packages/bluepy
make
Bluepy-helper not being built · Issue #158 · IanHarvey/bluepy · GitHub
So.. I did a fresh install of raspbian (debian stretch). I have installed python3-pip (pip3 version 9.0.1) install of bluepy (pip3 install bluepy) Then, when i run my program (that uses bluepy), i had trouble to access the device (a TI CC2650) So, i went in /usr/local/lib/python3.5/dist-packages/bluepy i run make some compilations are done then, i restart my program : it works well. Cheers, Sylvain
飛ばしてみる
$ python demoTricks

便利関数。これでだいたい飛ばせるYO。
def fly_direct(self, roll, pitch, yaw, vertical_movement, duration): """ Direct fly commands using PCMD. Each argument ranges from -100 to 100. Numbers outside that are clipped to that range. Note that the xml refers to gaz, which is apparently french for vertical movements: http://forum.developer.parrot.com/t/terminology-of-gaz/3146 :param roll: :param pitch: :param yaw: :param vertical_movement: :return: """ my_roll = self._ensure_fly_command_in_range(roll) my_pitch = self._ensure_fly_command_in_range(pitch) my_yaw = self._ensure_fly_command_in_range(yaw) my_vertical = self._ensure_fly_command_in_range(vertical_movement) command_tuple = self._get_command_tuple("Piloting", "PCMD") start_time = time.time() while (time.time() - start_time < duration): self.characteristic_send_counter['SEND_NO_ACK'] = (self.characteristic_send_counter['SEND_NO_ACK'] + 1) % 256 packet = struct.pack("<BBBBBBBbbbbI", self.data_types['DATA_NO_ACK'], self.characteristic_send_counter['SEND_NO_ACK'], command_tuple[0], command_tuple[1], command_tuple[2], 0, 1, my_roll, my_pitch, my_yaw, my_vertical, 0) self._safe_ble_write(characteristic=self.send_characteristics['SEND_NO_ACK'], packet=packet) #self.send_characteristics['SEND_NO_ACK'].write(packet) notify = self.drone.waitForNotifications(0.1)
今後
楽しみ♪
PARROT MAMBO FPV | Parrot Store Official
参考:
ノンプログラマでもコピペでOK!JavaScriptを使ってDroneを飛ばそう | 飛んでるあいつ
いまアツいJavaScript!ゼロから始めるNode.js入門〜5分で環境構築編〜
GitHub - sandeepmistry/noble: A Node.js BLE (Bluetooth Low Energy) central module
機械学習のお勉強(chainerでkaggleに初挑戦)
Kaggleとは
公式:Kaggle: Your Home for Data Science
Kaggleは企業や研究者がデータを投稿し、世界中の統計家やデータ分析家がその最適モデルを競い合う、予測モデリング及び分析手法関連プラットフォーム及びその運営会社である。
Kaggleを始めるのに参考になるサイト
Kaggle に Python で入門しました(Digit Recognizer) | Futurismo
Kaggle の Titanic チュートリアルに挑戦した話
Kaggleのtitanic問題で上位10%に入るまでのデータ解析と所感 - mirandora.commirandora.com
全体の流れ
Kaggle: Your Home for Data Science
アカウントを作る
トライするCompetitionを決める
Dataをダウンロードする
Dataを解析して、提出用csvファイルを作る
提出する
Digit Recognizerをやってみる(chainerを使用)
trainとtestのcsvファイルのダウンロード
chainer用のデータセットの作り方
とてもわかりやすい 👇
chainerのデータセットの作り方 LinearやCNN - Qiita
chainerの独自datasetの作り方 - 午睡二時四十分
ChainerのCNNにおけるデータセットの作り方 – ハックマン.com
Chainer: ビギナー向けチュートリアル Vol.1 - Qiita
chainerはtupleで[data,label]となるようにdatasetsを作ってから、学習を行う
Import
# -*- coding: utf-8 -*- import chainer import pandas as pd import chainer.functions as F import chainer.links as L from chainer import training from chainer.training import extensions import pandas as pd import numpy as np from chainer import datasets from chainer.datasets import tuple_dataset from chainer import serializers
kaggle dataをchainerデータセットに変換
train = pd.read_csv('./train.csv') test = pd.read_csv('./test.csv') features = train.ix[:,1:].values labels = train.ix[:,0] features = features.reshape(features.shape[0],28,28) imageData = [] labelData = [] for i in range(len(labels)): img = features[i] imgData = np.asarray(np.float32(img)/255.0) imgData = np.asarray([imgData]) imageData.append(imgData) labelData.append(np.int32(labels[i])) threshold = np.int32(len(imageData)/8*7) train = tuple_dataset.TupleDataset(imageData[0:threshold], labelData[0:threshold]) val = tuple_dataset.TupleDataset(imageData[threshold:], labelData[threshold:])
Trainerで学習
モデル
class CNN(chainer.Chain): def __init__(self, train=True): super(CNN, self).__init__( conv1=L.Convolution2D(1, 32, 5), conv2=L.Convolution2D(32, 64, 5), l1=L.Linear(1024, 10), ) self.train = train def __call__(self, x): h = F.max_pooling_2d(F.relu(self.conv1(x)), 2) h = F.max_pooling_2d(F.relu(self.conv2(h)), 2) return self.l1(h)
学習
model = L.Classifier(CNN()) optimizer = chainer.optimizers.Adam() optimizer.setup(model) train_iter = chainer.iterators.SerialIterator(train, batch_size=100) test_iter = chainer.iterators.SerialIterator(val, batch_size=100, repeat=False, shuffle=False) updater = training.StandardUpdater(train_iter, optimizer) trainer = training.Trainer(updater, (5, 'epoch'), out='result') trainer.extend(extensions.Evaluator(test_iter, model)) trainer.extend(extensions.LogReport()) trainer.extend(extensions.PrintReport( ['epoch', 'main/loss', 'validation/main/loss', 'main/accuracy','validation/main/accuracy'])) trainer.extend(extensions.ProgressBar()) trainer.run() serializers.save_npz("digit-learn5.data", model)
テストデータの準備
test = test.ix[:,:].values.astype('float32') test_data = test.reshape(test.shape[0],28,28) testData = [] for i in range(len(test_data)): img = test_data[i] imgData = np.asarray(np.float32(img)/255.0) imgData = np.asarray([imgData]) testData.append(imgData)
作成モデルでの予測
def predict(model, test):
# テストデータ全部に対して予測を行う
preds = []
for img in test:
img = img.reshape(-1, 1, 28, 28)
pred = F.softmax(model.predictor(img)).data.argmax()
preds.append(pred)
return preds
predictions = predict(model, testData)
kaggle提出用フォーマットに変換
submissions = pd.DataFrame({"ImageId": list(range(1, len(predictions)+1)),
"Label": predictions})
submissions.to_csv("digit_cnn_output.csv", index=False, header=True)
提出
ここのsubmit predictionsに上記でできたdigit_cnn_output.csvをアップロード
結果
527位 (/ 1661)

これからコツコツ学んだことを活かして、100位以内を目指してみる〜
コードも適当なので、もっと綺麗にかけるよう練習する
2017/10/11
331位

全体参考
nocotan.github.io 👈 最高にわかりやすい
GitHub - nocotan/chainer-examples: This is implementation examples by Chainer.
Chainer: ビギナー向けチュートリアル Vol.1 - Qiita
top2%の私が教えるKaggleの極意, Bosch Production Line Performance | RCO Ad-Tech Lab Blog
機械学習のお勉強(scikit-learnを使ってみる:分類問題)
教科書
Perceptron
実装
# -*- coding: utf-8 -* from sklearn import datasets import numpy as np # Added version check for recent scikit-learn 0.18 checks from distutils.version import LooseVersion as Version from sklearn import __version__ as sklearn_version if Version(sklearn_version) < '0.18': from sklearn.cross_validation import train_test_split else: from sklearn.model_selection import train_test_split # 正規化 from sklearn.preprocessing import StandardScaler # パーセプトロン from sklearn.linear_model import Perceptron from sklearn.metrics import accuracy_score # load data iris = datasets.load_iris() X = iris.data[:, [2, 3]] y = iris.target print('Class labels:', np.unique(y)) X_train, X_test, y_train, y_test = train_test_split( X, y, test_size=0.3, random_state=0) # 正規化 sc = StandardScaler() sc.fit(X_train) X_train_std = sc.transform(X_train) X_test_std = sc.transform(X_test) # Perceptron Fit ppn = Perceptron(n_iter=40, eta0=0.1, random_state=0) ppn.fit(X_train_std, y_train) # show accuracy y_pred = ppn.predict(X_test_std) print('Misclassified samples: %d' % (y_test != y_pred).sum()) print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
大事なのは
# Perceptron Fit ppn = Perceptron(n_iter=40, eta0=0.1, random_state=0) ppn.fit(X_train_std, y_train)
結果
Misclassified samples: 4 Accuracy: 0.91
結果を可視化(コードは教科書Git参照) 👇

3クラス分類において、線形の決定境界で完全に区切ることができない
ロジスティック回帰
分類のためのモデルで、線形分類問題と2値分類問題に使用される

ロジスティック関数
起こりやすさを表すオッズ比(P =p/(1-p))の対数の逆関数

入力として実数を受け取り、[0,1]の範囲の値に変換する。出力はそのクラスに所属している確率と解釈できる。
例として、降水確率や病気の確率を出す際などに利用されている
コスト関数
J = cost func p = probability if y==0: J = -log(1-p) elis y==1: J = -log(p)

実装
上記パーセプトロンのPerceptron Fit以降を下のように書き換える
from sklearn.linear_model import LogisticRegression lr = LogisticRegression(C=1000.0, random_state=0) lr.fit(X_train_std, y_train) y_pred = lr.predict(X_test_std) print('Misclassified samples: %d' % (y_test != y_pred).sum()) print('Accuracy: %.2f' % accuracy_score(y_test, y_pred)) # output probability print lr.predict_proba(X_test_std[0,:])
結果
Misclassified samples: 1 Accuracy: 0.98 [[ 2.05743774e-11 6.31620264e-02 9.36837974e-01]]
パーセプトロンより向上している

lr = LogisticRegression(C=1000.0, random_state=0)
のCについて以下で説明する
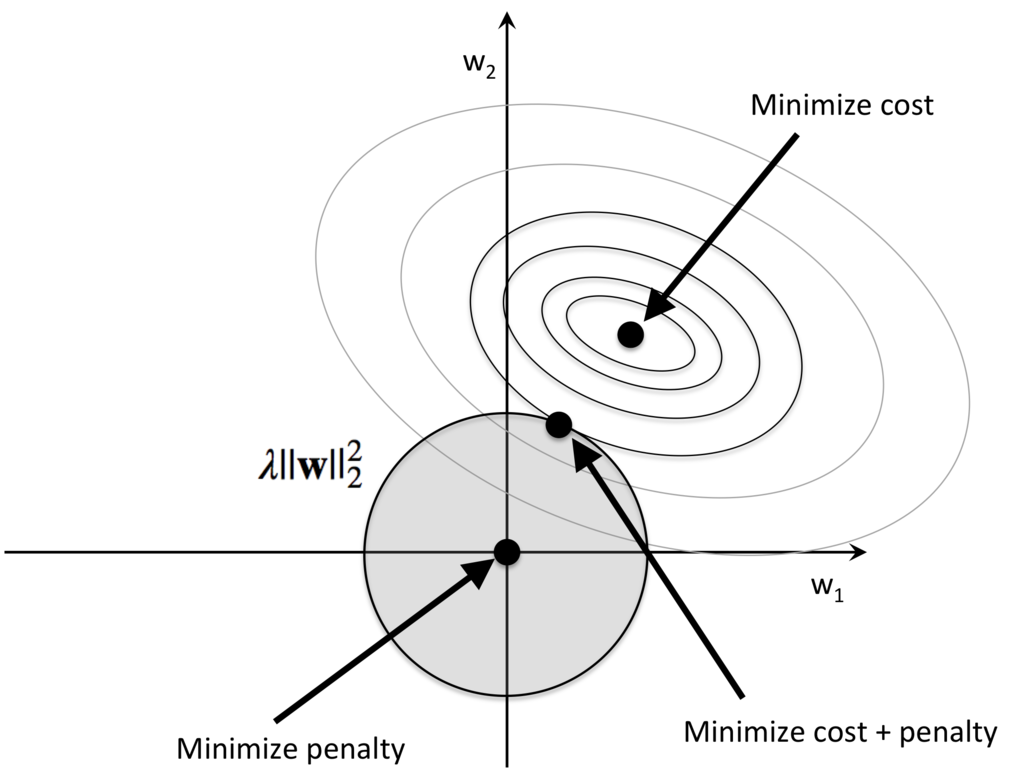
L2正規化
極端なパラメータの重みにペナルティをかす。コスト関数に正規化の項を追加すればよい
+ lambda / 2 * w2 **をコスト関数に足すことで、さらには、lambdaを大きくすることで、重みを小さく保ちながら学習させることができる。
J = cost func p = probability w = weight if y==0: J = -log(1-p) + lambda / 2 * w**2 elis y==1: J = -log(p) + lambda / 2 * w**2
C = 1 / lambdaを表している。Cの値を減らすと重みが小さくなる。

正規化をするメリットは?
直感的には、正規化することによりニューラルネットワークがより小さな重みを好むようになります。 大きな重みが許されるのは、そうすることがコスト関数の第1項を余程大きく改善する場合だけです。 言い換えると、正規化とは重みを小さくすることと元のコスト関数を小さくすることの間でバランスを取る方法であると 見ることもできます。このバランスを取る上で、2つの要素の相対的な重要性を決定するのが λλ の値です: λλ が小さい時は元のコスト関数を最小化することを好み、λλ が大きい時には より小さな重みを好むのです。
さて、実際のところ、そのようなバランスを取ることがなぜ過適合を軽減する助けになるのか、 その理由は誰の目にもすぐさま明らかなものではありません。しかし、正規化が実際に過適合を軽減することは分かっています。
もっと深堀りすると 👇
正規化されたニューラルネットワークで期待されるように、 ニューラルネットワークの大部分では小さな重みを持つと仮定しましょう。 重みが小さいということは、ここそこでランダムな入力を変化させても ニューラルネットワークの振る舞いが大きくは変わらないことを意味します。 そのため、正規化されたニューラルネットワークでは、 データに含まれる局所的なノイズの効果を学習しづらくなっています。 その代わり、正規化されたニューラルネットワークは訓練データの中で繰り返し見られる データの特徴に反応するのです。対照的に、大きな重みを持つニューラルネットワークは、 入力の小さな変化に敏感に反応してその振る舞いを大きく変えてしまいます。 そのため、正規化されていないニューラルネットワークは、大きな重みを使って、 訓練データのノイズに関する情報をたくさん含んだ複雑なモデルを学習してしまうのです。 要するに、正規化されたニューラルネットワークは訓練データに頻繁に現れるパターンに基づいた 比較的シンプルなモデルを構築します。そして、訓練データが持つノイズの特異性を学ぶことに対して 耐性を持つのです。このため、ニューラルネットワークがノイズではなく現象そのものに対する真の学習をして、 それをより良く汎化できるのではないかと、希望が持てます。
サポートベクターマシン
マージン最大化を行う

コスト関数
cost func = 1 / 2 * w**2
スラック変数を用いた際のコスト関数
epsilon = slack variable cost func = 1 / 2 * w**2 + C * (sum(epsilon))
このCを変化させることで、バイアスとバリアンスのトレードオフを調整できる

二次計画法
数学的に考えてみる(ハードマージンSVM) - Shogo Computing Laboratory
http://numaf.net/Z9/Z9a/html/THESIS/H15/abst_toda.pdf
http://www.r.dl.itc.u-tokyo.ac.jp/~nakagawa/SML1/kernel1.pdf
実装
上記パーセプトロンのPerceptron Fit以降を下のように書き換える
svm = SVC(kernel='linear', C=1.0, random_state=0) svm.fit(X_train_std, y_train) y_pred = svm.predict(X_test_std) print('Misclassified samples: %d' % (y_test != y_pred).sum()) print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
結果
Misclassified samples: 1 Accuracy: 0.98

カーネルSVM
考え方:射影関数によってデータを高次元空間へ射影し、線形分離できるようにする

デメリット
射影変換(phi(x))を行うと本来、学習過程で生じるxxである計算がphi(x)phi(x)となるため、計算コストがかかる
しかし、カーネルトリックを用いることで軽減できる
k(x_i,x_j) = phi(x_i) * phi(x_j) # Radial Basis Function kernel k(x_i,x_j) = exp(-(x_i - x_j)**2 / (2 * sigma**2)) # Simple Radial Basis Function kernel k(x_i,x_j) = exp(- gamma *(x_i - x_j)**2 )
Radial Basis Function kernelは2つのサンプル間の類似度を表してる
実装
上記パーセプトロンのPerceptron Fit以降を下のように書き換える
先のkernelのパラメータがlinearからrbfに変わっただけ 👇
svm = SVC(kernel='rbf', random_state=0, gamma=0.10, C=10.0) svm.fit(X_train_std, y_train) y_pred = svm.predict(X_test_std) print('Misclassified samples: %d' % (y_test != y_pred).sum()) print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
パラメータのgammaは
# Simple Radial Basis Function kernel k(x_i,x_j) = exp(- gamma *(x_i - x_j)**2 )
のgammaで小さくするとなめらかに、大きくすると複雑になる
gamma = 0.2にすると 👇

gamma = 100.0にすると 👇

未知のデータでは汎化誤差が生じることが予想される
決定木
意味解釈可能性に配慮する場合に良いモデル

上記のようなカテゴリだけでなく実数でも可能。閾値を1.0などと決めるだけで良い。
決定木の深さが深くなる場合、深さの制限や剪定が必要になる。
情報利得の最大化
目的関数(二分木)
親ノードの不純度と子ノードの不純度の合計の差。つまり、子ノードの不純度が低いほど情報利得は大きくなる
I = Impurity(不純度) Dp = Parent DataSet Dleft = Child DataSet Dright f = function I(Dp, f) = I(Dp) - Nleft/Np * I (Dleft) - Nright/Np * I (Dright)
不純度:エントロピー
t:ノード
相互情報量が最大化になるよう試みる。p(1|t)=1 or p(0|t)=0の時エントロピーは0。p(1|t)=1/2 or p(0|t)=1/2の時エントロピーは1。
Ih = - sum(p(i|t)log2 p(i|t))
不純度:ジニ不純度
誤分類の確率を最小化
Ig = sum(p(i|t) * (1 - p(i|t))) = 1 - sum(p(i|t)**2)
エントロピーと同様、p(1|t)=1 or p(0|t)=0の時エントロピーは0。p(1|t)=1/2 or p(0|t)=1/2の時エントロピーは1。
不純度:分類誤差
すべてのサンプルが最大条件付き確率を与えるクラスに所属すると予測した時に間違えるサンプルの割合を表現する
Ie = 1 - max(p(i|t))
不純度の比較

実装
上記パーセプトロンのPerceptron Fit以降を下のように書き換える
決定木の場合、特徴量のスケーリングは必要ない。
from sklearn.tree import DecisionTreeClassifier tree = DecisionTreeClassifier(criterion='entropy', max_depth=3, random_state=0) tree.fit(X_train, y_train) y_pred = tree.predict(X_test_std) print('Misclassified samples: %d' % (y_test != y_pred).sum()) print('Accuracy: %.2f' % accuracy_score(y_test, y_pred)) # 決定木の可視化 from sklearn.tree import export_graphviz export_graphviz(tree, out_file='tree.dot', feature_names=['petal length', 'petal width'])


ランダムフォレスト
決定木のアンサンブルと考えることができる
トレーニングデータからランダムにn個のサンプルを抽出
決定木を成長させる
上記を繰り返す
決定木ごとの予測をまとめて多数決に基づいてクラスラベルを割り当てる
「はじめてでもわかる RandomForest 入門-集団学習による分類・予測 -」 -第7回データマイニング+WEB勉強会@東京
メリット
パラメータの設定に悩む必要が少なく、上記ステップ1,2を何回繰り返すかを設定すれば良い。繰り返す回数を増やせば性能が上がるが計算コストが増える。
ポイント
上記ステップ1のnは多くの場合サンプルと同じ数に設定するのが良い
実装
上記パーセプトロンのPerceptron Fit以降を下のように書き換える
10個決定木からランダムフォレストをトレーニング
from sklearn.ensemble import RandomForestClassifier forest = RandomForestClassifier(criterion='entropy', n_estimators=10, random_state=1, n_jobs=2) forest.fit(X_train, y_train) y_pred = forest.predict(X_test_std) print('Misclassified samples: %d' % (y_test != y_pred).sum()) print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))

k近傍法
怠惰学習の代表例。識別関数を学習せずトレーニングデータセットを暗記する。ノンパラメトリックモデル。
kの値と距離指標を選択
分類したいサンプルからk個の最近傍のデータ点を見つける
多数決によりクラスラベルを割り当てる

実装
from sklearn.neighbors import KNeighborsClassifier knn = KNeighborsClassifier(n_neighbors=5, p=2, metric='minkowski') # Power parameter for the Minkowski metric knn.fit(X_train_std, y_train) y_pred = knn.predict(X_test_std) print('Misclassified samples: %d' % (y_test != y_pred).sum()) print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))

結果
Misclassified samples: 0 Accuracy: 1.00